
格式化DNA柱状图 |

|

|

|
|
|
格式化DNA柱状图 |

|

|

|
格式化DNA柱状图
|
|
||
FCS Express中有很多格式化功能,它们帮助用户对多周期DNA柱状图分析做一个视觉上的评估。不同的细胞周期细胞群可以有不同的阴影,用户可以显示某一细胞群中间值对应的标记。另外,用户可以在柱状图上对某一区域进行放大,以便更细致地查看绘图。
首先让我们在多周期柱状图上把二倍体细胞群的颜色修改为单色。
| 1. | 请点击多周期柱状图将其选中。 |
| 2. | 请选中Format(格式)→Plot Options(绘图选项)→Overlays(叠加图层)命令。 |
这时会出现Formatting Overlays(格式化叠加图层)对话框,如图T19.23所示。以下步骤均针对图T19.23。
| 3. | 请从左边选中Diploid G1(二倍体G1),如图中的蓝色所示。 |
| 4. | 请从下拉菜单中的Fill Style(填充样式)中选择单色,如图中的光标所示。 |
| 5. | 请点击OK。 |
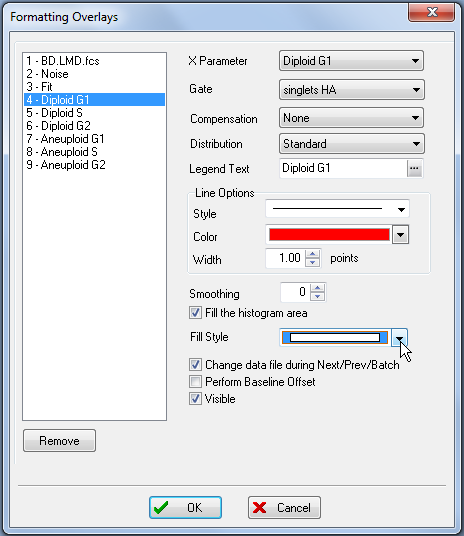
图T19.23 Formatting Overlays Dialog for a MultiCycle DNA Histogram (多周期DNA柱状图的格式化叠加图层对话框)
这时多周期柱状图会进行更新,反映出图T19.24中进行的修改。或者,我们可以右键点击DNA柱状图,调出弹出菜单,选择Format(格式),然后选择Overlays(叠加图层)来对柱状图叠加图层进行格式化。
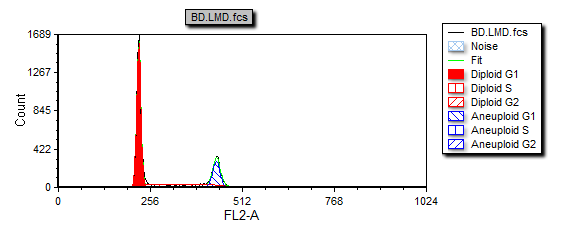
图T19.24 MultiCycle DNA Histogram Updated with Solid Fill on the Diploid Peak (更新后的多周期DNA柱状图中的单色二倍体数据峰)
现在我们要在柱状图上添加一个标记,表明非整倍体G2峰值的位置。
| 6. | 请右键点击DNA柱状图,调出弹出菜单。 |
| 7. | 请从菜单上选择DNA Position Markers(DNA位置标识)→A2 Mean(A2平均值),如图T19.25中的光标所示。 |
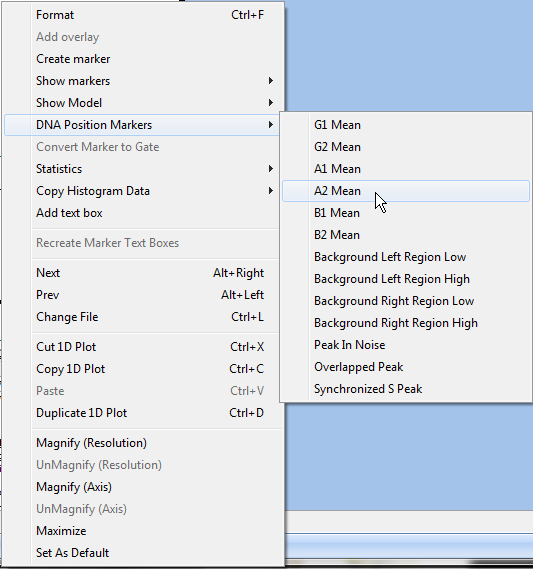
图T19.25 Selecting DNA Position Markers on the Pop-up Menu (从弹出菜单上选择DNA位置标识)
或者,DNA位置标识可从弹出菜单上的Format(格式)一项来调出。
| • | 请右键点击柱状图,调出弹出菜单。 |
| • | 请从菜单是选中Format(格式)。 |
这时会出现柱状图格式化窗口,以下步骤均针对图T19.26。
| • | 请从左边的类别选项选中DNA→Position Markers(位置标识),如图中的淡蓝色显示。 |
| • | 请选中Show position marker on the plot(在绘图上显示位置标记)复选框,如图中的光标所示。 |
| • | 请选中A2 Mean(A2平均值),如图中的蓝色高亮所示。 |
| • | 请点击OK。 |
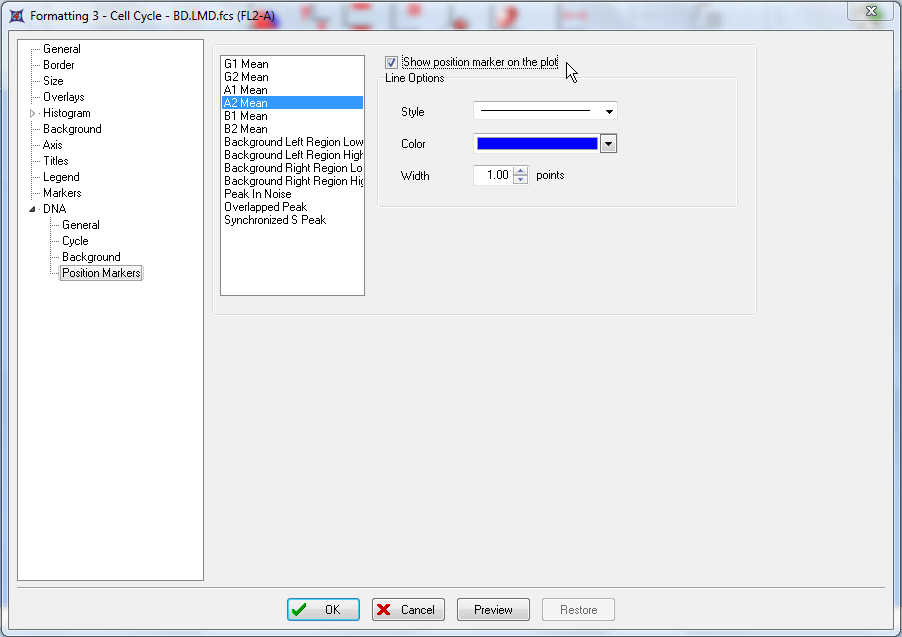
图T19.26 Formatting DNA Histogram Position Markers (格式化DNA柱状图位置标识)
这时在多周期DNA柱状图上非整倍体G2信号峰的平均值位置处会出现一条垂直线(图T19.27)。
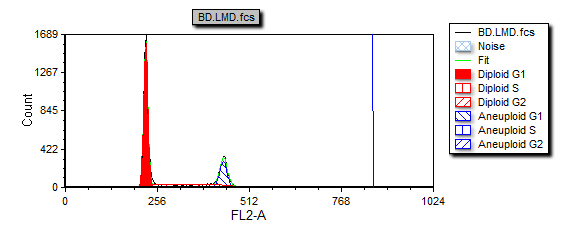
图T19.27 MultiCycle Histogram Updated with a DNA Position Marker on the Aneuploid G2 Mean (更新过的多周期柱状图以及代表非整倍体G2平均数的DNA位置标识)
我们现在要在DNA柱状图上进行放大,来更近地考察一下非整倍体细胞群。
| 8. | 请右键点击DNA柱状图,调出弹出菜单。 |
| 9. | 请从弹出菜单上选中Magnify (Axis)(放大(轴))(图T19.25)。 |
以下步骤均针对图T19.28。
| 10. | 请把光标放在DNA柱状图上非整倍体G1数据峰的左侧。 |
| 11. | 请按下鼠标左键向右移。 |
| 12. | 当向右的箭头在DNA位置标识的右侧时,请松开鼠标(如图中的光标所示)。 |
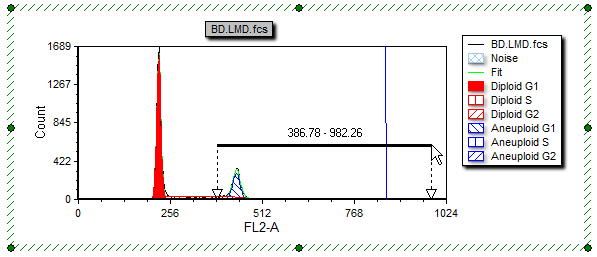
图T19.28 Selecting an Area on the X-axis to Zoom (在X-轴上选中一个区域来进行放大)
柱状图这时已经进行了更新,放大到前几步选中的x-轴部分,如图T19.29所示。
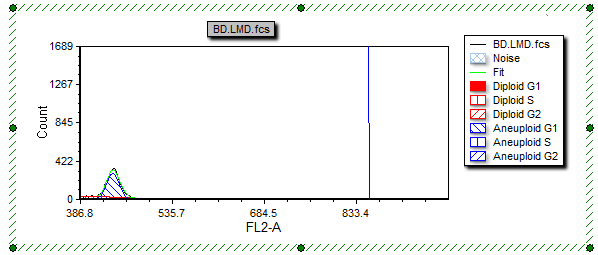
图T19.29 Histogram Zoomed (放大后的柱状图)
我们现在要修改柱状图上的y-向坐标。
| 13. | 请点击柱状图,将其选中。 |
| 14. | 请选中Format(格式)→Plot Options(绘图选项)→Axes(坐标轴)命令。 |
这时会出现Formatting Axes(格式化坐标轴)对话框。以下步骤请参考图T19.30。
| 15. | 请选中Y-Axis(Y-轴),如图中的蓝色高亮所示。 |
| 16. | 请点击Automatic(自动)旁边的复选框,取消对该选项的选择,如图中的光标所示。 |
| 17. | 请输入"300",作为Y-轴的最大值,如图中的蓝色高亮所示。 |
| 18. | 点击OK。 |
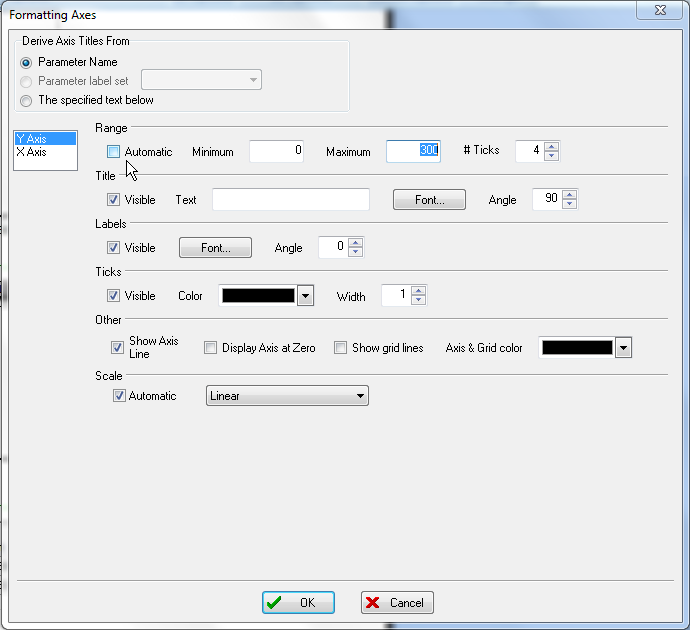
图T19.30 Formatting Axes Dialog (格式化坐标轴对话框)
这时多周期柱状图会进行更新,y-轴的范围变成0到300,如图T19.31所示。
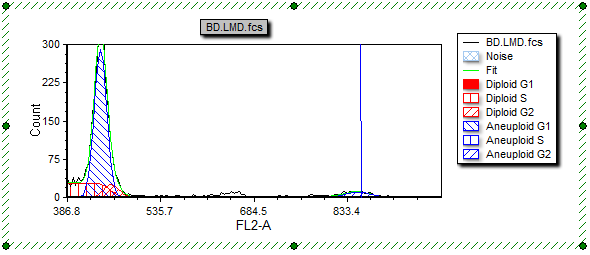
图T19.31 MultiCycle Histogram with Rescaled Y-axis (多周期柱状图以及调整后的Y-轴)
在下一节中,我们将对3周期数据使用多周期分析。