DNA周期和模型摘要参考
FCS Express中的MultiCycle AV(简易型多周期软件包)能够自动拟合适当的周期来匹配用户的数据。但是,用户可以不用自动拟合功能,而是自己选择一个周期来匹配您的数据。
这里我们对MultiCycle AV(简易型多周期软件包)中不同周期做了简介。
1 Cycle(单周期) |
它是最普通也是最常用的分析。当有一个完整的细胞周期存在时,就使用它。 |
1 Cycle Standard Overlay Peak(单周期标准重叠峰) |
与1 Cycle(单周期)类似。当使用一个标准化工具时,如鸡红细胞,就使用它。 |
1 Cycle Apoptotic Overlay Peak(单周期凋亡重叠峰) |
当假定在G0/G1峰的左边有一个额外的峰,就使用这种周期分析方法。 |
1 Cycle Aneuploid Overlay Peak(单周期非整倍体重叠峰) |
当有一个非整倍体峰存在,而不包含一个完整的细胞周期,就使用这种周期分析方法。 |
1 Cycle Diploid Overlay Peak(单周期二倍体重叠峰) |
当有一个二倍体峰存在,而不包含一个完整的细胞周期,就使用这种周期分析方法。 |
2 Cycle(两周期) |
当有2个完整的细胞周期存在时,就使用它。 |
3 Cycle(3周期) |
当有3个完整的细胞周期存在时,就使用它。 |
Synchronous S Phase(同步S期) |
当实验中S被中断,导致S期有一个峰,而不是平稳渐进曲线,这种情况下,就使用Synchronous S Phase(同步S期)周期分析方法。 |
FCS Express的MultiCycle AV(简易型多周期软件包)可在同一时间,应用6种不同的模型到您的数据中。
在FCS Express中,您可以创建一个DNA Model Statistics Summary box(DNA模型统计摘要框),来显示应用到这6种不同模型中的统计。
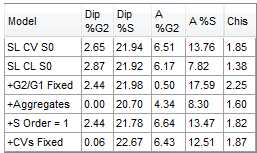
DNA Model Statistics
这里我们对MultiCycle AV(简易型多周期软件包)中不同周期做了简介。
SL S0 |
是最常用的模型。带有zero order S phase(0顺序S期)的切核背景模型,但不带有其他选项。当碎片曲线看上去像滑雪斜坡时,请使用它。 |
SL CL S0 |
带有聚集补偿和zero order S phase(0顺序S期)的切核背景模型。如果碎片曲线是平的且在G0/G1期中切断,请使用它。 |
+G2/G1 Fixed |
添加了G2/G1比率限制的切核背景模型。对于单周期拟合模型,G2/G1比率是当前默认储存在MultiCycle AV(简易型多周期软件包)中的数据。
请注意:对于2周期模型,二倍体周期的G2/G1比率被强制等于非整倍体G2/G1比率,它来自用户选择的拟合模型。
对于3周期模型,3周期的G2/G1比率被强制等于两个非整倍体G2/G1比率,它来自用户选择的拟合模型。 |
+Aggregates(聚集) |
添加了聚集模型和G2/G1比率约束的切核背景模型。可以说,这是一个具有双重辨别的文件版本。这个模型将计算所有可能的G1和G2粘连,然后输出粘连细胞的百分比。 |
+S Order = 1 |
应用了第一顺序S期多项式的切核背景模型。当S期的形状扩大为梯形时,请使用该分析方法。
S order = 0 (broadened rectangle)(S顺序= 0(扩大为矩形)) S order = 1 (broadened trapezoid)(S顺序=1(扩大为梯形)) S order = 2 (higher on the sides, lower in the middle)(S顺序=2(两边更高,中间更低)) |
+CVs Fixed |
所有峰添加了CVs约束的切核背景模型。当G2/M峰埋入S期中时,请使用该周期分析。 |
要了解更多详细信息,请参考“多周期知识页面”章节。
如果数据不是最佳的或者没有定义好细胞周期峰,用户可以得到Intra-model(内部-模型)或者Intra-model(内部-模型)错误信息。关于此错误信息的定义,您可在以下网址中找到:http://www.denovosoftware.com/site/kb-DNAMulticycleInfo.shtml。
