Analysis of 3 Cycle Data(分析3周期数据)
对包括多周期细胞群的DNA柱状图的分析需要多周期软件包高级版本,该版本需要另行购买。首先我们将打开一个空白版面页,插入一个有多个非整倍体细胞群的DNA柱状图。
| 1. | 选择File(文件)标签→New(新的可用模板)→New(创建一个新的空白版面). |
| 2. | 请选择Insert(插入)标签→1D Plots(一维绘图)→MultiCycle DNA(多周期DNA)命令。 |
| 3. | 请在版面上的空白区点击。 |
此Select a Data File(选择一数据文件)对话框将出现,如前图T19.1所示.
| 4. | 请导航到FCS Express Sample Data(FCS Express样本数据)文件夹下。 |
| 5. | 选择文件ASCII4.fcs. |
| 6. | 点击Open(打开). |
这时会出现多周期DNA柱状图,如图T19.33所示。注意到图上的两个非整倍体细胞群(分别显示为蓝色以及绿色),占据了柱状图中绝大部分空间。
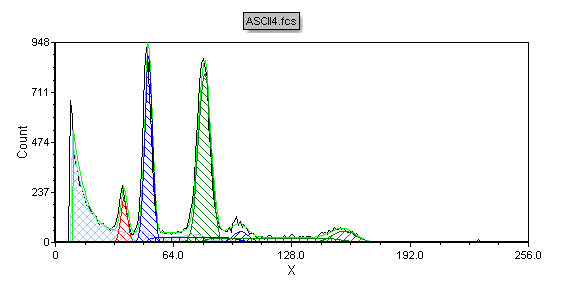
Figure T19.33 MultiCycle DNA Histogram with Two Aneuploid Populations
用户可以插入DNA Cycle(DNA周期),DNA Model Summary(DNA模型摘要), 和DNA Experiment Statistics(DNA实验统计)窗口(如前几节演示的那样)来进一步研究该柱状图。但很明显这个DNA柱状图有多周期细胞群,多周期软件包似乎也正确的识别了它们。
我们现在来看一个更难分析的柱状图,因为非整倍体细胞群相互交叠且多周期软件包中的自动拟合功能也未能正确地锁定这两个非整倍体细胞群,因此我们需要手动进行设置。首先让我们来插入柱状图。
| 7. | Select theInsert(插入)标签→1D Plots(一维绘图)→Histogram(柱状图)命令。 |
| 8. | 请在版面上的空白区点击。 |
一个当前数据文件的柱状图将出现在版面中。要修改绘图上的数据,请按以下操作:
| 9. | 右击柱状图,然后从弹出菜单选择Change file (变更文件)选项。 |
| 10. | 选择文件ASCII8.fcs从出现的Select a Data File(选择一数据文件)对话框中。 |
| 11. | 点击Open(打开). |
此时,这一柱状图将显示ASCII8.fcs文件的数据,见图T19.34。注意,图中有一个非整倍体数据峰,如图中的红圈所示。实际上,仔细观察该图就会发现图中有两个DNA含量相似的非整倍体数据峰。我们现在将使用多周期软件包的自动拟合功能来分析该DNA柱状图,并且演示针对这一情况,这一分析不会给出正确结果。
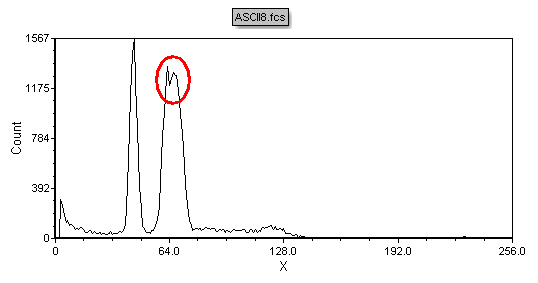
Figure T19.34 ASCII8.fcs Histogram
| 12. | 选择Insert(插入)标签→1D Plots(一维绘图)→MultiCycle DNA(多周期DNA)命令(T19.1所示). |
| 13. | 请在版面上的空白区点击。 |
此Select a Data File(选择一数据文件)对话框将出现,如前图T19.1所示.
这时会出现多周期DNA柱状图,如图T19.33所示。多周期软件包的自动拟合功能已经使用两周期模型对柱状图进行了分析,并认为图中只有一个非整倍体细胞群,如图中的蓝色所示。我们现在要在不运用自动拟合的情况下来分析DNA柱状图。
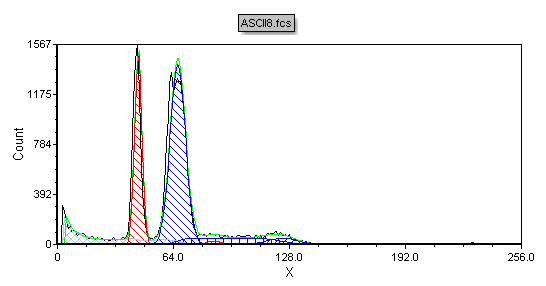
Figure T19.35 Autofit MultiCycle Analysis of ASCII8.fcs
| 17. | 请在ASCII8.fcs对应的多周期柱状图上点击,将其选中。 |
| 18. | 选择Multicycle(多周期)→Fit Parameters(拟合参数)命令。 |
此Formatting Fit Model Options(格式化拟合模型选项)对话框出现。接下来步骤请参见图T19.36。
| 19. | 选择取消对话框 |
| 20. | 选择下拉菜单中 |
| 21. | 选中 |
| 22. | 点击OK. |
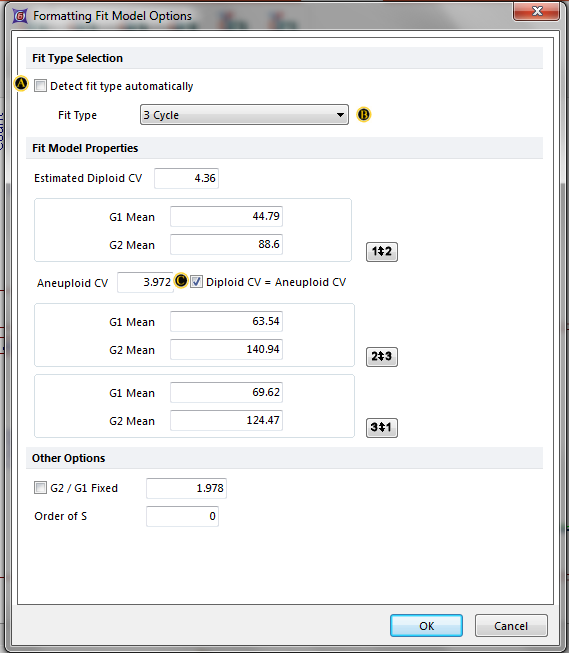
Figure T19.36 Formatting Fit Model Options Dialog
现在,我们已经关闭了自动拟合功能并设置多周期软件包使用3周期模型.同时,我们也对该模型的设置了附件限制:二倍体信号峰的CV应该和非整倍体信号峰的CV相同。这时,在柱状图上会显示使用手动设置进行的多周期拟合,如图T19.37所示。
另外,用户可以在Multicycle(多周期)标签卡上关闭自动拟合功能,然后选中3周期模型。
| • | Select theMulticycle(多周期)→Fit Parameters(拟合参数)→Autofit(自动拟合)命令,来关闭自动拟合功能。 |
| • | 选择Multicycle(多周期)→Fit Parameters(拟合参数)→3 Cycle(3周期)从周期下拉列表选择 |
| • | 请右击ASCII8多周期柱状图,调出弹出菜单。 |
| • | 选择Format(格式)从弹出菜单中。 |
此Formatting Cell Cycle(格式化细胞周期)窗口将出现。
| • | 从对话框左侧选择DNA→Cell Cycle(细胞周期)类别选项。 |
此DNA 细胞周期对话框中的细胞周期格式出现,外观和Formatting Fit Model Options(拟合模型选项格式)相似,见图T19.36 。
| • | 选中 |
| • | 点击OK. |
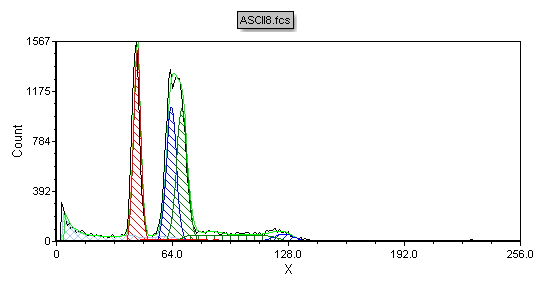
Figure T19.37 Manual MultiCycle Fit
