自动拟合的扩展分析
现在我们将要调整单峰HA,圈出包含更高DNA含量的细胞。在基本版本中的多周期自动拟合会继续执行单周期拟合。但是,高级版本中的多周期自动拟合会精确地确定,是应用一个、二个或者三个周期去匹配直方数据。
门的最终位置展示于图T19.16。
| 1. | 点击门内部选中“单峰”门编辑。 |
| 2. | 移动光标到顶部右上角调整门。当光标移到正确位置时,光标会变成一个两向的箭头。 |
| 3. | 当向上拖动门一角时,按下鼠标左键。 |
| 4. | 当把门一角拖到所需的位置时,请松开鼠标(图T19.16)。 |
| 5. | 重复步骤2到4调整门的顶部左上角。 |
门如图19.6所示,修饰含有更高DNA含量的细胞
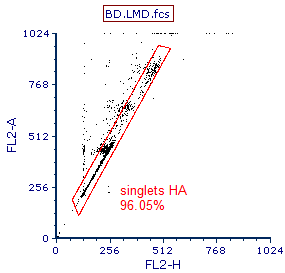
Figure T19.16 Gate Modified to Include Cells with Higher DNA Content
多周期的自动拟合功能更新了细胞周期分析,如图T19.17所示。注意:图T19.17显示了使用高级版本中的多周期来进行多周期分析。在基本版本中的多周期拟合会继续应用单周期模型到直方图中。
在本教程剩下的部分,我们将使用高级版本中的多周期来进行DNA分析。但是,DNA柱状图的所有的格式化功能和相关的数据在基础版本上也是可用的,并且可以用相同的方式进行分析。
注意DNA拟合现在会显示非整倍体群体,如图19.17中蓝色所示。T事实上,多周期解释说明这个最像一个四倍体群体。同时也需要注意也应用到了二周期模型。在实验数据窗口会显示这个信息,并且在选择DNA柱状图和选择多周期标签时也可以发现。Multicycle(多周期)标签。
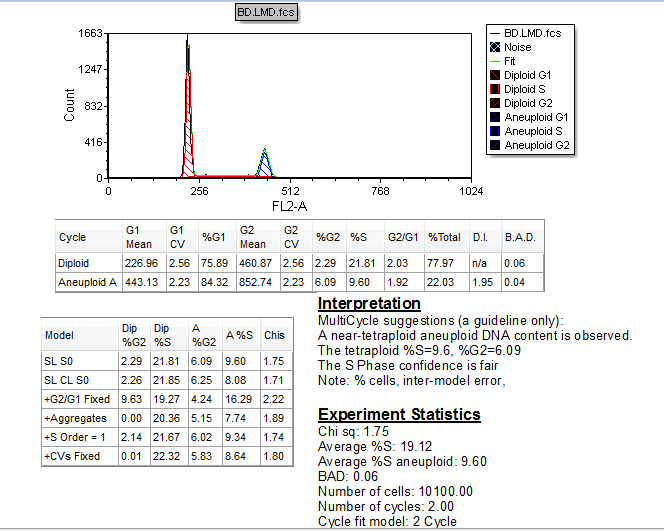
Figure T19.17 Autofit MultiCycle Analysis with Modified gate
现在我们将选择我们所需要的具体的数据DNA Model Summary Statistics(DNA模型摘要统计)表格有多种方式选择表中所显示的统计数据,并且这些相同的方法可以用来选择在弹出菜单中显示为任意DNA相关联的数据。图T19.13要更多地了解“格式化”内容,请参考本教程格式化绘图首先,我们会关闭DNA Experiment Statistics(DNA实验统计)窗口,这样就会有空间去扩大DNA Model Summary Statistics(DNA模型摘要统计)窗口。
| 6. | 请把光标移到边界DNA Experiment Statistics(DNA实验统计)窗口。 |
| 7. | 当光标变成四方向箭头时,点击鼠标左键,边界会变成红色,表明你选中并且可编辑。 |
| 8. | 点击Del键;第二种方法是,选择Home(主页)→Editing(编辑)。 |
| 9. | 点击鼠标左键DNA Model Summary Statistics(DNA模型摘要统计)窗口去选择它。 |
| 10. | 选择Format(格式)→Statistics Options(统计选项)→Statistics(统计)命令。 |
此格式化数据选项对话框将出现(图T19.18).
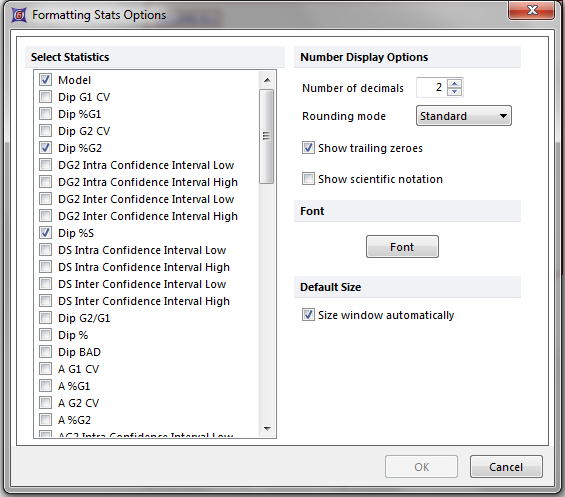
Figure T19.18 Formatting Stats Options Dialog for DNA Model Summary Statistics
| 11. | 选择下接箭头,如T19.18中的光标所示。 |
| 12. | 请不要松开鼠标左键,向下滚动到列表的底部。 |
| 13. | 点击旁边的框簇和碎片如图T19.19所示。 |
| 14. | 点击OK。 |
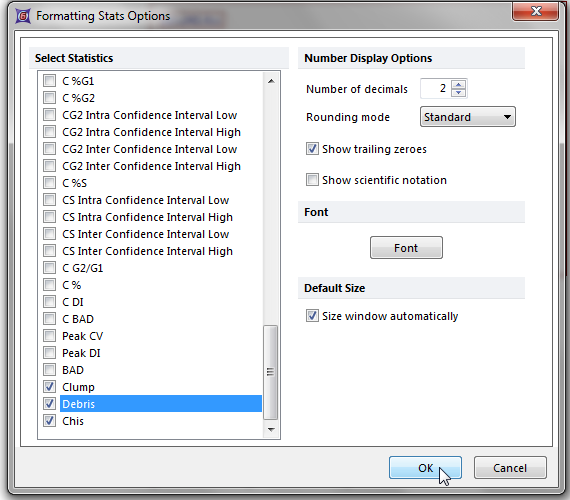
Figure T19.19 Selecting Clump and Debris from the Formatting Stats Options Dialog
此DNA Model Summary Statistics(DNA模型摘要统计)表格会更新,并显示选择的统计数据,如图T19.20所示。
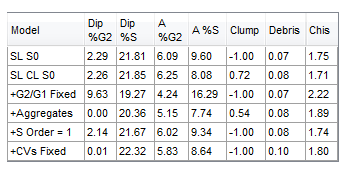
Figure T19.20 DNA Model Summary Statistics Table Updated to Show to Clump and Debris
或者,用户可在工具栏使用DNA Model Summary Statistics(DNA模型摘要统计)调出相关的弹出菜单,或者取消选择,具体统计数据显示
| • | 请右键点击DNA Model Summary Statistics(DNA模型摘要统计)窗口的顶端边线处,来调出相关的弹出菜单。 |
此DNA Model Summary Statistics(DNA模型摘要统计)出现弹出菜单(图T19.21)。
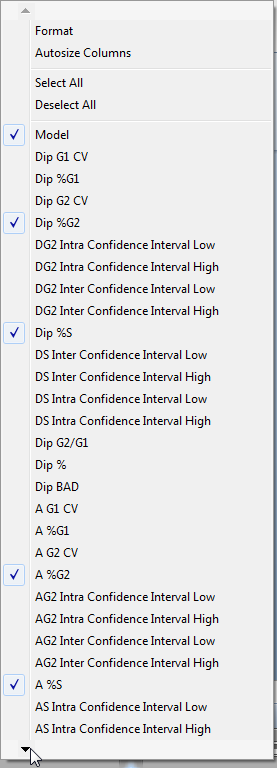
Figure T19.21 DNA Model Summary Statistics Pop-up Menu
| • | 选择左下角的箭头向下滚动菜单,如图T19.21所示。 |
| • | 请不要松开鼠标左键,直到菜单卷轴的最底端(图T19.22所示)。 |
| • | 点击鼠标左键旁边的簇,如图T19.22中的光标所示,取消选定该项。 |
关闭弹出式菜单,然后DNA Model Summary Statistics(DNA模型摘要统计)表格会更新,来反映该项的移除簇。
| • | 重复以上步骤,取消选定碎片,位于簇如图T19.22。 |
关闭弹出式菜单,然后DNA Model Summary Statistics(DNA模型摘要统计)表格会更新,来反映该项的移除碎片。
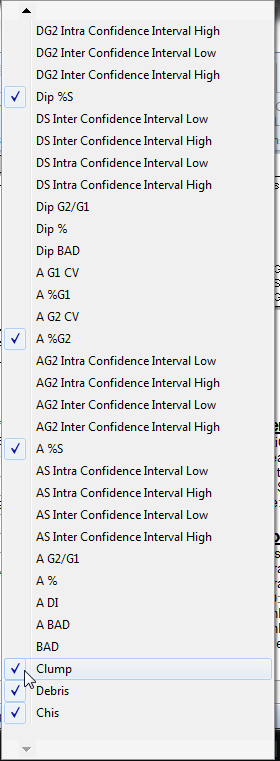
Figure T19.22 Deselecting Clump from the DNA Model Summary Statistics Pop-up Menu
用户可以选择不同的DNA细胞周期模型Multicycle(多周期)→→Model to Display(要显示的模型)组,并且DNA细胞周期数据窗口会更新,反映出选择的模型。尝试选择不同的模型Multicycle(多周期)标签自动拟合在DNA细胞周期数据窗口可以观察到结果是如何变化的。当使用多周期完整版本的多周期自动拟合,直方图显示也会更新,反映选择的模型是否适合。不同的模型通过其复杂性变化(应用约束,或者不是,CV值或者G2/G1平均值比率)要想更多地了解“不同的模型”,请参考多周期。
一个DNA模型的快速视觉检查通常来说都比较有用。但是,卡方检验值最低的模型Chis(卡方检验)值最低的通常是最合适的,如图T19.23中用红色圆圈标记部分。在本例中,模型2(SL CL S0)具有最低的Chis;但是,其它两个模型具有非常相似的低Chis值。
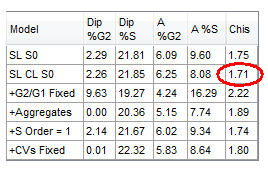
Figure T19.23 DNA Cell Cycle Model with the Lowest Chis (Chi-square) Value
