选择自动拟合(单周期分析)
对门进行设置来排除重叠的细胞:
要排除成对的细胞,我们将检查DNA荧光信号的高度与DNA荧光面积信号的二维绘图,然后在单个细胞事件上画门。很多时候用户也会使用DNA荧光信号的宽度与DNA荧光信号的面积之间的二维绘图。信号的高度对应于从DNA染料发出的荧光信号的峰值。面积是DNA染料发出的荧光信号曲线下的计算面积。信号宽度代表DNA染料发出的荧光脉冲的时间。具体的激光束和其它设备条件将决定哪些参数最适合把单细胞与重叠的细胞区分开来。在我们的例子中,DNA荧光面积信号定义的参数是FL2-A,DNA荧光宽度信号对应的参数是FL2-W,DNA荧光高度对应的参数是FL2-H。
如果已经打开一个空白版面了,请跳到第2步。
| 1. | 请选择File(文件)标签→New(新的可用模板)→New(创建一个新的空白版面)。 |
| 2. | 请选择Insert(插入)标签→2D Plots(二维绘图)→Dot(点状图)命令。 |
| 3. | 请在空白版面内任何地方点击。 |
此时,Select a Data File(选择一数据文件)对话框将出现(图T19.1)。
| 4. | 请来到TutorialSampleData文件夹。 |
| 5. | 请确保选中的文件类型是FCS files(FCS文件)(*.*, *.fcs, *.lmd)格式。 |
| 6. | 请选择BD.LMD.FCS文件(图T19.1所示)。 |
| 7. | 点击Open(打开)。 |
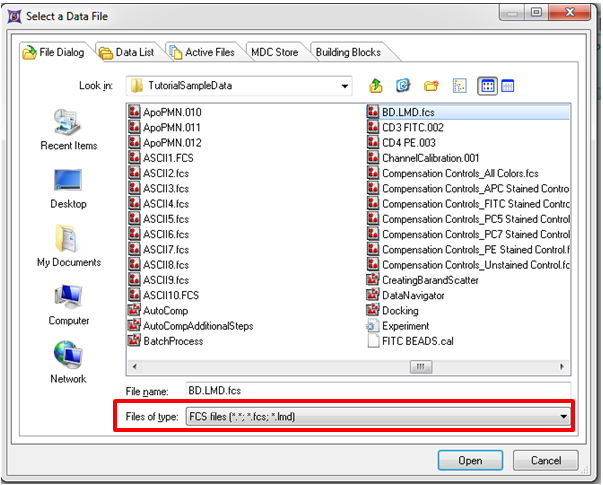
Figure T19.1 Select a Data File Dialog
这时版面上会出现一个点状图。
| 7. | 请点击x-轴,然后从弹出菜单上选择FL2-H选项。 |
| 8. | 请点击y-轴,然后从弹出的参数菜单上选择FL2-A选项。这时点状图应该看起来如图T19.2所示。 |
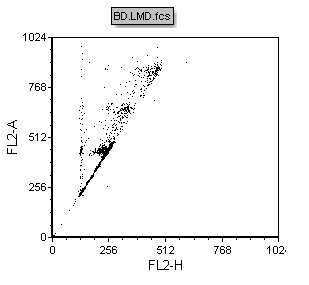
Figure T19.2 Dot Plot of FL2-H vs FL2-A
我们现在要在二维绘图上创建一个门来排除粘在一起的两个细胞以及那些看起来像是四倍体DNA的数据。因为我们想要执行的是单循环DNA分析,所以要排除那些有高含量DNA的细胞。以下步骤展示于图T19.3。想要更多的了解如何创建门,请参考门操作教程。
| 9. | 请选择Gating(门操作)标签→Create Gates(创建门)→Polygon(多边形门)命令。 |
| 10. | 创建一个如图T19.3中所示的多边形门。 |
此时,Create a New Gate(创建一个新门)对话框将出现。
| 12. | 请把门命名为"singlets HA"。 |
这时该门应该看起来如图T19.3所示。
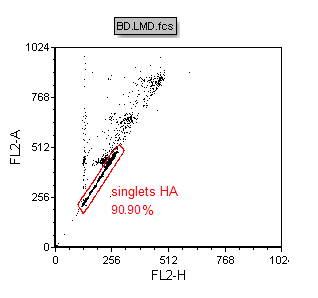
Figure T19.3 Gate to Exclude Doublets and Cells iwth High DNA Content
我们现在已经可以使用多周期软件包中的自动拟合功能来执行DNA细胞周期分析了。
插入多周期柱状图:
| 13. | 请选择Insert(插入)标签→1D Plots(一维绘图)→MultiCycle DNA(多周期DNA)命令(图T19.4)。 |
| 14. | 请在版面上的空白区点击。 |
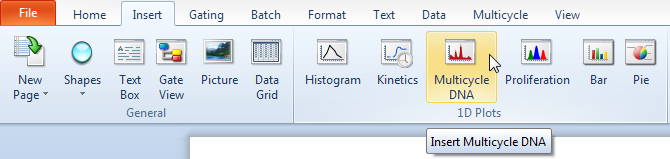
Figure T19.4 Selecting the Insert Muticycle DNA Command
此时,Select DNA Parameter(选择DNA参数)对话框将出现,如图T19.5所示。以下步骤均展示于图T19.5。
| 18. | 请选择FL2-A作为参数,如图中的蓝色高亮所示。 |
| 19. | 点击OK。 |
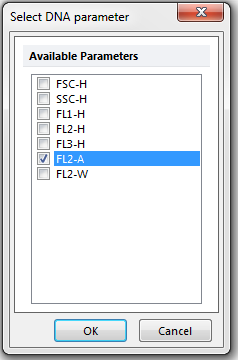
Figure T19.5 Select DNA Parameter Dialog
应用singlets HA门细胞的FL2-A柱状图经过使用自动拟合的多周期分析,如图T19.6所示。注意:由于圈门的形状有差别,因此您绘图上的数据可能和图T19.6显示的有所不同。
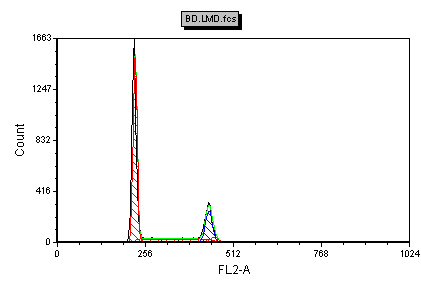
Figure T19.6 MultiCycle Analysis of FL2-A Using Singlets HA Gate
我们将把感兴趣的多周期绘图区域放大,用以提高适应度。
| 20. | 右键点击Multicycle plot(多周期绘图)然后从弹出菜单中选择Magnify resolution(放大分辨率)选项。 |
| 21. | 按住鼠标左键不动,将光标拖拽(此时光标是十字符号光标)到您感兴趣的位置(图T19.7所示)。 |
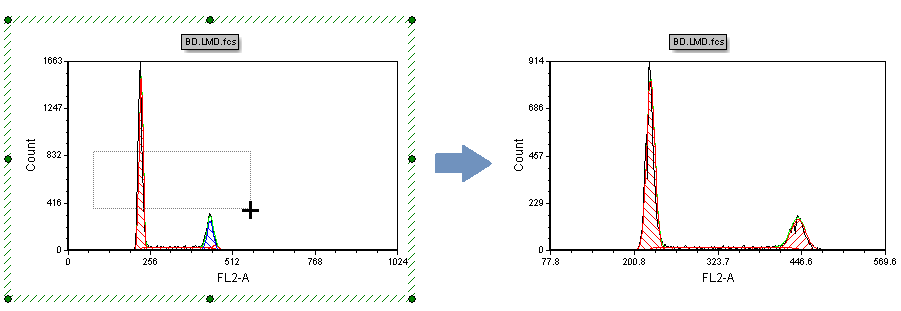
Figure T19.7 Magnifying resolution on a MultiCycle plot to improve the fit of the model
| 22. | 然后松开光标。此多周期绘图的拟合已经更新,然后现在只有一个群体被检测,已用红色表示出来(图T 19.7所示)。 |
我们现在将显示和多周期DNA分析相关的图例信息。
| 21. | 请右键点击柱状图,调出相关弹出菜单。 |
| 22. | 请从弹出菜单中选择Format(格式)选项。 |
| 23. | 请从Formatting(格式化)对话框中分类中选择Legend(图例)选项(图T19.8)。 |
| 24. | 点击Visible(可视)复选框。 |
| 25. | 点击OK(图T19.8)。 |
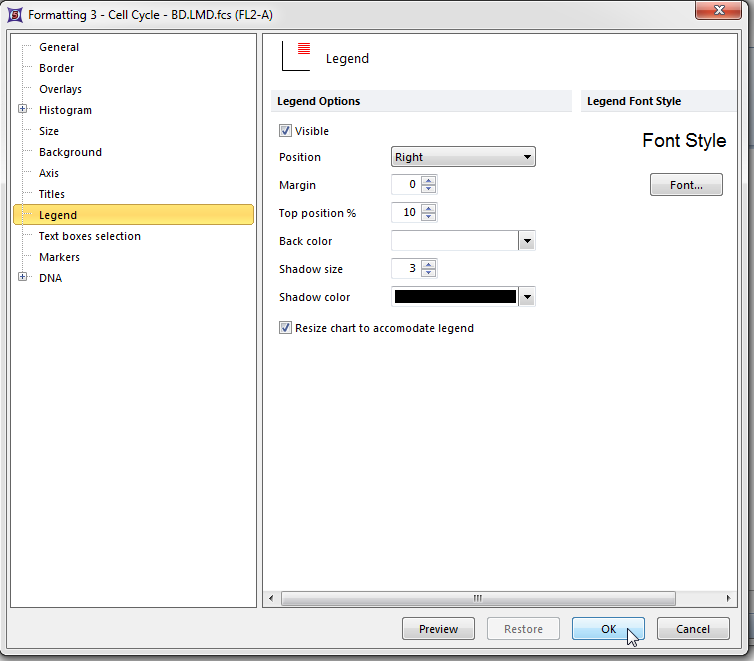
Figure T19.8 Formatting the DNA histogram to Display the Legend Associated with MultiCycle Analysis
或者,可用Format Legend(格式化图例)命令来显示图例信息,方法如下:
| • | 请单击柱状图,柱状图应该有一个绿色边框。 |
请选择Format(格式)→Plot Options(绘图选项)→Legend(图例)命令(图T19.9)。
Figure T19.9 Selecting the Format Legend Command
此时,Formatting Legend(格式化图例)对话框将出现(图T19.10)。
| • | 请在Formatting Legend(格式化图例)对话框中选中Visible(可视)复选框。 |
| • | 点击OK。 |
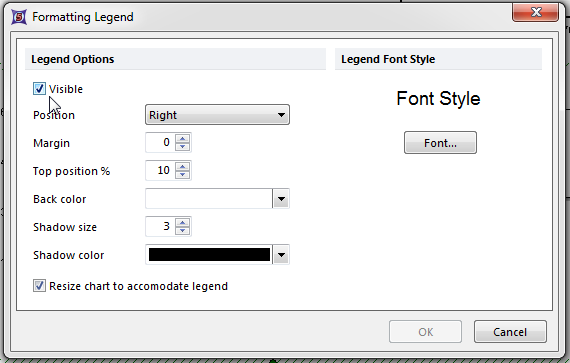
Figure T19.10 Formatting Legend Dialog
这时和多周期DNA分析相关的图例会出现在柱状图的右侧(图T19.11)。
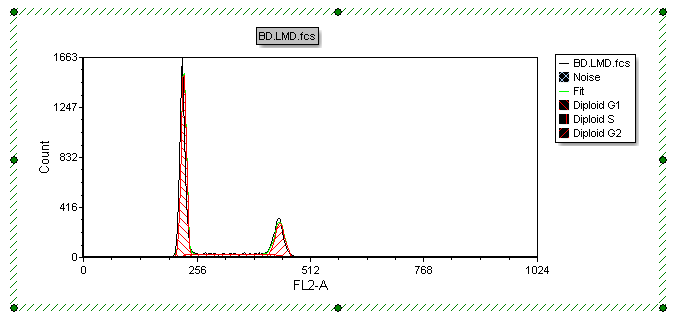
Figure T19.11 MultiCycle DNA Histogram Cell Cycle Analysis with Associated Legend
如你在图例中看到的那样,这个柱状图仅仅包括二倍体细胞,因此可以用多周期软件包的自动拟合功能来执行单周期分析。我们可以通过检测Ribbon条上的多周期信息来确认这一点。
| 26. | 请左键点击柱状图将其选中,这时柱状图应该有一个绿色边框(图T19.10)。 |
| 27. | 请选择Multicycle(多周期)标签(图T19.12)。 |
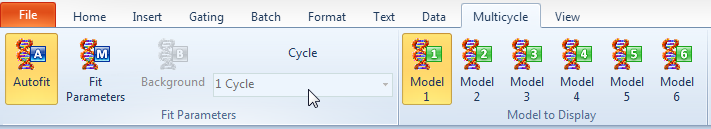
Figure T19.12 The Multicycle Tab
注意:此时Autofit(自动拟合)和Model 1 (模型1)被高亮显示为橙色(图T19.12)。这表明多周期拟合模型和模型1正被用来分析这个DNA柱状图。同时也注意到图T19.11中光标所指的单周期也被用来进行分析。
注:要想了解不同模型之间的区别,请参考De Novo Software DNA/多周期知识章节。
我们现在要来查看这个柱状图的所有针对DNA分析的不同统计。
| 28. | 请右键点击柱状图,调出相关弹出菜单。 |
| 29. | 请从弹出菜单中选择Statistics(统计)→DNA Cycle Statistics(DNA周期统计)(图T19.13)。 |
Figure T19.13 DNA Cycle Statistics Pop-up Menu Item
当前模型Model 1的DNA Cycle Statistics(DNA周期统计)窗口将出现(图T19.14)。注意:用户可能有和在这里看到的不同的统计数值,因为用户的单细胞门可能和这节中的门不同。
| 30. | 移动该DNA Cycle Statistics(DNA周期统计)窗口倒绘图的下方(图T19.14)。 |
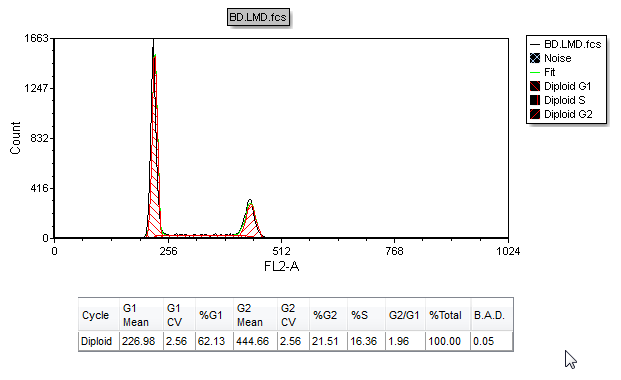
Figure T19.14 DNA Cell Cycle Statistics from the MultiCycle Model 1 Analysis of the Histogram
| 31. | 请右键点击DNA 柱状图,调出相关弹出菜单。 |
| 32. | 请从弹出菜单中选择Statistics(统计)→DNA Model Summary Statistics(DNA模型摘要统计)如前图T19.13所示。 |
此时,DNA Model Summary Statistics(DNA模型摘要统计)窗口将出现,它显示了所有应用于当前柱状图的定义好的多周期模型,共六个。此时,DNA Model Summary Statistics(DNA模型摘要统计)窗口中的六个模型分别对应于Multicycle(多周期)→Model to Display(要显示的模型)组中的六个模型。(图T19.12)。在FCS Express中,DNA Model Summary Statistics(DNA模型摘要统计)显示的默认统计参数设置,稍后的教程中我们将修改对其进行修改。
| 33. | 移动该DNA Model Summary Statistics(DNA模型摘要统计)窗口到DNA Cell Cycle Statistics(DNA细胞周期统计)窗口下方(图T19.15)。 |
| 34. | 请右键点击DNA 柱状图,调出相关弹出菜单。 |
| 35. | 请从弹出菜单中选择Statistics(统计)→DNA Experiment Statistics(DNA实验统计)如前图T19.13所示。 |
此时,DNA Experiment Statistics(DNA实验统计)窗口将出现,窗口中有对DNA模型的解释以及实验统计数据。
| 36. | 移动该DNA Experiment Statistics(DNA实验统计)窗口到DNA Model Summary Statistics(DNA模型摘要统计)窗口下方(图T19.15)。 |
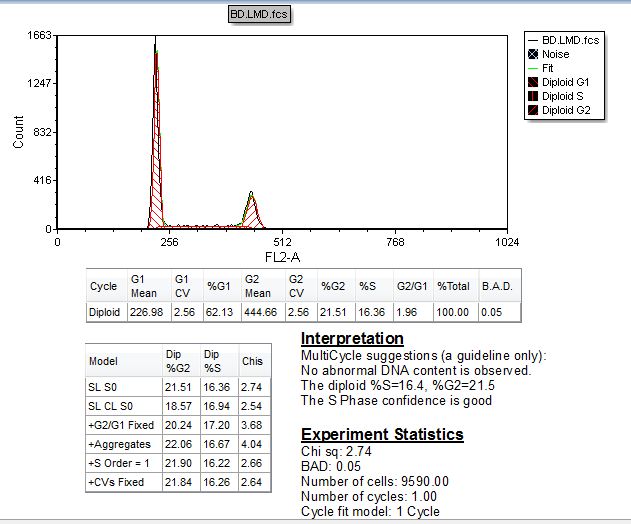
Figure T19.15 DNA Histogram with All of the MultiCycle Statistics Windows
在下一节中,我们将介绍自动拟合的扩展分析。
