导出来自CellProfiler的大平铺图片
导出大平铺图片
大对象或者大样本量的高分辨率图片,通常是通过将很多小的图片平铺来完成的(通常称为一拼接图片)。这些图片通常是由采集仪器或者软件通过以下两种方式提供。通常,它们会自动拼接在一起,然后提供出一张大的图片。这种情况下,它们必须被打散成小的图片,以供CellProfiler分析,然后FCS Express会把它们重新装配成一大图片。或者,它们以多个小图片的方式提供。这种情况,CellProfiler就可以直接分析这些小图片,然后FCS Express会把它们重新装配成一大图片(可选操作)。
在本教程中,我们将介绍:
| • | FCS Express所需的图片命名规范,从而让CellProfiler产生的平铺数据能够导入FCS Express中。 |
| • | 在CellProfiler中,如何将一大图片分隔成多个平铺图片。 |
| • | 从CellProfiler中导出平铺图片,以供在FCS Express中使用 |
平铺图片的命名规范
当重新拼接一系列的平铺图片时,FCS Express会使用保存在文件名称中的信息,来将图片正确的拼接在一起。尤其是后面跟有编号的"row"和 "col"信息,表明了平铺图片在大图片中的位置。下面是关于这一命名规范的例子:
mytiledimage_row00_col00.tif
mytiledimage_row00_col01.tif
mytiledimage_row00_col02.tif
上面的三个文件名称代表前三个平铺小图片,这可由它们的"row"和"col"位置来证明。如果可能,在获取图片的同时,请为平铺图片添加"row"和"col"字样及相关的位置;或者,在获取后,手动为文件重命名或者使用一个重命名程序。
以大图片开始
可用Cell Profiler和FCS Express对大图片进行分析。由于很多电脑的储存限制,这些图片必须被分割为多个小图片。我们将以使用Total Image Slicer(总图片切片机)将大图片分割为500 x 500像素的正方形小图片,设置文件名称模板为:[name]_row%row_col%col.tiff ([名称]_行%行_列%列.tiff)。(总图片切片机将同时且自动的将图片切割为分散的正方形,并且用"rowXX" "colXX"命名规范对它们进行重命名)。
从CellProfiler中导出平铺图片,以供在FCS Express中使用
本节中将讨论的CellProfiler模块包括:Load Images(导入图像)、Convert Objects to Images(把对象转化为图像)、Save Images(保存图像)以及Export to Spreadsheet(导出到工作表)。本例中设置好的管道文件可以在Tutorial Sample Data archive(教程样本数据库)中的Exporting Tiled Images in CP(导出平铺图片到电脑)文件夹下找到,名称是CellProfilerTiledExportCOMPLETED.cp。这个设置好的、作为模板的管道可以用来和用户的管道进行比较,且代表本教程的成品。
本例中,我们将使用的示例数据集可以在Tutorial Sample Data archive(教程样本数据库)中的Exporting Tiled Images in CP(导出平铺图片到电脑)文件夹下找到,名称是“In_Situ_Cerebellum_Staining”。此示例数据集(kindly provided by Dragan Maric, Ph.D(由Dragan Maric博士友情提供)) 表示一个3色+光亮区域实验,该实验中,一大鼠的小脑被成像。要访问管道的最终产品和数据的一个版面,请参见Applications Examples webpage(应用样本网页)。
从CellProfiler导出数据的步骤已经根据CellProfilerTiledExport.cp管道模块进行了分解。在CellProfiler中导入CellProfilerTiledExport.cp管道后,请遵循以下步骤来修改管道,以便为向FCS Express导出做准备。
选择默认的输入和输出文件夹
要为FCS Express正确地组织数据,首先请保证在CellProfiler中默认的输入(存储图片的地方)以及输出文件夹是同一个文件夹(在Input/Output Folder(输入/输出文件夹)窗口中进行设置)(图T24.43)。
注意:DefaultOUT.mat文件也将被导出到Output Folder(输出文件夹)。这个文件可在MATLAB中使用。如果您不想用这个文件,我们建议将其删除,以节省硬盘空间。
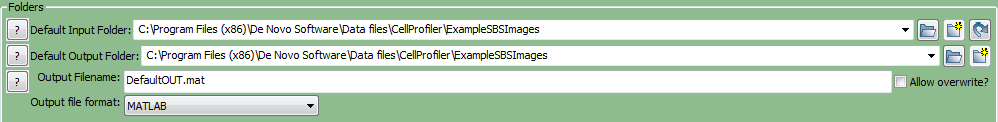
Figure T24.43 Set the Default Input and Output Folders to the Same Location
设置LoadImages Module(导入图片模块)
要想让FCS Express读出平铺图片的以行和列形式表示的数据,图片文件名中的元数据必须被提取出来。必须定义一Regular Expression(一般表达式)来找到文件名或数据路径中的元数据,方法请遵照以下的步骤(图T24.44):
| 1. | 点击DAPI-下拉列表标签Extract metadata from where?(从哪里提取元数据?)。 |
| 2. | 请选择你想要使用的元数据。对本例来说,我们选择File name(文件名称)。 |
| 3. | 请在Regular expression that finds metadata in the file name(在文件名中寻找元数据的一般表达式)字段中输入你想要使用的"Regular Expression(一般表达式)"。在本例中,我们使用“_0000_350_DAPI_row(?P<rowtile>[0-9]{1,3})_col(?P<coltile>[0-9]{1,3}).tiff”,该表达式定义在图片名称中找到的、每个平铺图片的行和列信息。 |
注意:
| • | 请点击以下链接来查看"Regular Expression(一般表达式)"教程以及“如何翻译一般表达式”教程。 |
| • | 我们只需为数据集中的一个图片设置元数据以及一般表达式。在本例中,我们为DAPI通道做了这些设置。 |
| • | 一般表达式的输出名字:微孔、行、列可能只适用于基于96孔板或者拼接图像的实验。将这些名称用于任何单一的图像实验,将导致一个非-工作输出。 |
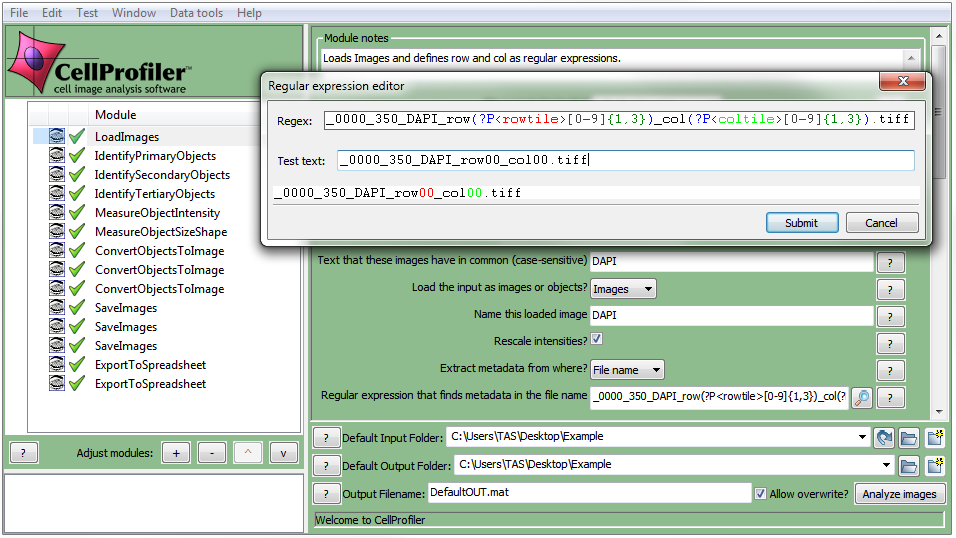
Figure T24.44 Setting up a Regular Expression in the LoadImages Module for a tiled image experiment.
ConvertObjectsToImage Modules (把对象转化为图片模块)
在该模块中,"Identify Primary/Secondary Objects(确认主要/次要对象)"模块中定义好的对象将被转化为FCS Express在导入和分析中可以使用的图片蒙版。图T24.45给出了一个该模块窗口的例子。
| 4. | 在管道中 选择 第一个ConvertObjectsToImage(把对象转化为图片)模 块。 |
| 5. | 从Select the input objects(选择输入对象)下拉列表中选择Nuclei(细胞核)。 |
| 6. | Name the output image(为输出图片命名)让用户输入想要的名称。在本例中,使用"Nuclei(细胞核)"(注意区分大小写)。 |
| 7. | 从下拉列表中选择颜色类型,然后选择“uint16”(图T24.3)。 |
| 8. | 在管道中选择第二个ConvertObjectsToImage(把对象转化为图片)。 |
| 9. | 从Select the input objects(选择输入对象)下拉列表中选择Cells(细胞)。 |
| 10. | Name the output image(为输出图片命名)让用户输入想要的名称。在本例中,使用" Cells(细胞)"(注意区分大小写)。 |
| 11. | 从下拉列表中选择颜色类型,然后选择“uint16”. |
| 12. | 在管道中选择第三个ConvertObjectsToImage(把对象转化为图片)。 |
| 13. | 从Select the input objects(选择输入对象)下拉列表中选择Cytoplasm(细胞质)。 |
| 14. | Name the output image(为输出图片命名)让用户输入想要的名称。在本例中,使用" Cytoplasm(细胞质)"(注意区分大小写)。 |
| 15. | 从下拉列表中选择颜色类型,然后选择“uint16”。 |
注意:在定义你自己的管道时,请为你通过Identify Primary/Secondary Objects(确认主要/次要对象)模块定义的对象重复第4步到第7步。
(注意:为了让FCS Express正确的输入Name the output image(为输出图片命名)数据,名称必须与Select the input objects(选择输入对象)名称一样,且注意区分大小写)
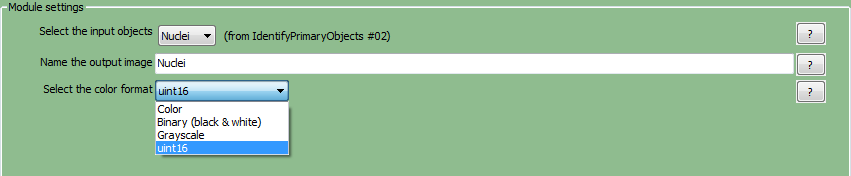
Figure T24.45 ConvertObjectsToImage Module: Defining Nuclei and Cells Image Masks
SaveImages Module (保存图片模块)
我们在ConvertObjectsToImage(把对象转化为图片)模块中定义的图像蒙版必须被保存下来,并且和原始的图像/数据进行关联。
| 12. | 在管道中 选择第一个SaveImages(保存图片)模块。 |
| 13. | 请在Select the type of image to save(选择保存的图片类型)下拉列表中选择Image(图片)。 |
| 14. | 将Select the image to save(选择要保存的图片)设置为Cells(细胞)。 |
注意:定义自己的管道时,请在ConvertObjectsToImage(把对象转化为图片)模块中,选择您定义的适当的图片。
| 15. | 请在Select method for constructing file names(选择构建文件名的方法)下拉列表中选择single name(单一名称)。 |
| 16. | 请在Enter single file name(输入单个文件名)字段输入任何你想要的名字,然后再输入一个连字符"_rowtile_columntile"。在本例中,我们使用CellLabel_rowtile_columntile。 |
| 17. | 请在CellLabel_rowtile后点击右键,(但要在"_"之前),选择rowtile(行瓦片)。 |
| 18. | 请在columntile后点击右键,然后选择coltile(列瓦片)。 |
| 19. | 在管道中 选择第二个SaveImages(保存图片)模块。 |
| 20. | 请在Select the type of image to save(选择图片保存类型)下拉列表中选择Image(图片)。 |
| 21. | 将Select the image to save(选择要保存的图片)设置为Nuclei(细胞核)。 |
| 22. | 请在Select method for constructing file names(选择构建文件名的方法)下拉列表中选择Single name(单一名称)。 |
| 23. | 请在Enter single file name(输入单个文件名)字段输入任何你想要的名字,然后再输入一个连字符"_rowtile_columntile"。在本例中,我们使用NucleiLabel_rowtile_columntile。 |
| 24. | 请在NucleiLabel_rowtile 后点击右键(但要在"_"之前),选择rowtile(行瓦片)。 |
| 25. | 请在columntile后点击右键,然后选择coltile(列瓦片)。 |
| 26. | 在管道中 选择第三个SaveImages(保存图片)模块。 |
| 27. | 请在Select the type of image to save(选择图片保存类型)下拉列表中选择Image(图片)。 |
| 28. | 将Select the image to save(选择要保存的图片)设置为Cytoplasm(细胞质)。 |
| 29. | 请在Select method for constructing file names(选择构建文件名的方法)下拉列表中选择Single name(单一名称)。 |
| 30. | 请在Enter single file name(输入单个文件名)字段输入任何你想要的名字,然后再输入一个连字符"_rowtile_columntile"。在本例中,我们使用CytoplasmLabel_rowtile_columntile。 |
| 31. | 请在CytoplasmLabel_rowtile后点击右键(但要在"_"之前),选择rowtile(行瓦片)。 |
| 32. | 请在columntile后点击右键,然后选择coltile(列瓦片)。 |
注意:rowtile(行瓦片)和coltile(列瓦片)是您为导入图片模块定义的一般表达式数值。使用一般表达式,允许根据输入文件名中的文本,为每个输出文件设置一个独一无二的名称。这将防止输出文件覆盖其它的输出文件,并让FCS Express可以找到每个图片文件对应的图片蒙版。
为所有针对CellLabel、NucleiLabel和 CytoplasmLabel设置的SaveImages (保存图片)模块执行第33-39步。
| 33. | 请从Select the format to use(选择使用的格式)下拉列表中选择tif。 |
| 34. | 在Image bit depth(图片比特深度)下拉列表中设置16。 |
| 35. | 从Output file location(输出文件位置)中选择Default Output Folder(默认输出文件夹)。 |
| 36. | 请选中Overwrite existing files without warning?(覆盖已有文件时不进行警告?)对话框。 |
| 37. | 在Select how often to save(选择如何保存)下拉列表中选择Every cycle(每个周期)。 |
| 38. | 请在Select colormap(选择颜色图)下拉列表中选择gray(灰色)。 |
| 39. | 请选中Update names within CellProfiler?(在CellProfiler里更新名称?)对话框。 |
此时,针对CellLabel的SaveImages(保存图片)模块看上去将如图T24.46所示。
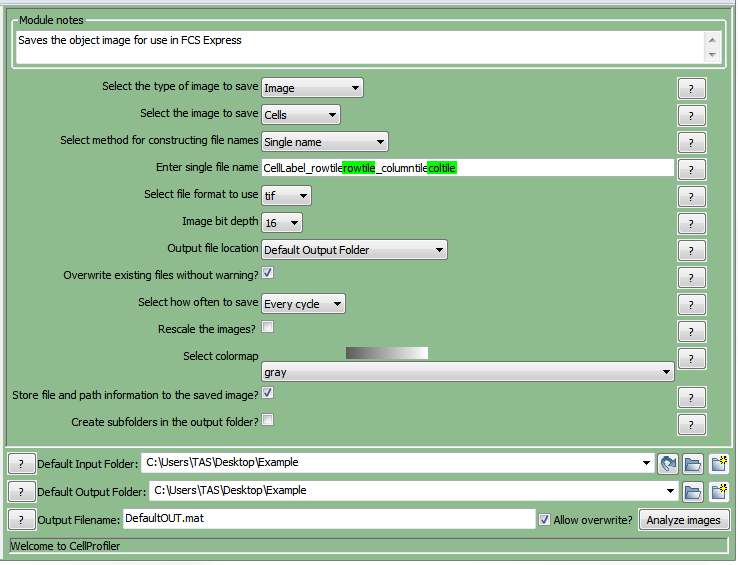
Figure T24.46 Defining the SaveImages Module
ExportToSpreadsheet Module (导出到电子表格模块)
既然所有的“图片”以及“对象图片蒙板”都已经定义并保存好了,用户现在即可让CellProfiler导出用户想要在FCS Express中查看的测量数据。在本例中,我们将导出选中的测量数据。
(注意:如果您只想要导出包含参数的数据,请参见“为CellProlifer导出选择特定参数”章节)
| 33. | 选中第一个ExportToSpreadsheet(导出到电子表格)模块。 |
| 34. | 请从下拉列表中选择Comma(逗号)(",")作为列分隔符。 |
| 35. | 取消对复选框Prepend the output file name to the data file names?(在数据文件名称前追加输出文件名?)的选择。 |
| 36. | 从Output file location(输出文件位置)下拉列表中选择Default Output Folder(默认输出文件夹)。 |
| 37. | 取消对Export all measurements, using default file names?(输出所有测量数据,使用默认文件名?)的选择。 |
| 38. | 从第一个Data to export(要导出的数据)下拉列表中选择Image(图片)。 |
| 39. | 取消对Use the object name for the file name(使用对象名作为文件名?)的选择。 |
| 40. | 请把文件名设置为Image.cptoc。 |
| 41. | 选择第二个ExportToSpreadsheet(导出到电子表格)模块。 |
| 42. | 请从下拉列表中选择或输入Comma(逗号)(",")作为列分隔符。 |
| 43. | 取消对Prepend the output file name to the data file names?(在数据文件名称前追加输出文件名?)对话框的选择。 |
| 44. | 从Output file location(输出文件位置)下拉列表中选择Default Output Folder(默认输出文件夹)。 |
| 45. | 选中复选框Select the columns of measurements to export?(选择测量值中的列进行输出)。 |
注意:在本例中,测量值已经被选中,nuclei.cpout文件也已经设置好。点击Press to select measurements button(按下选择测量值按钮)来检查哪种测量值被选中。
| 46. | 选择Add another data set(添加另外一组数据集)。 |
| 47. | 从第二个Data to export(要导出的数据)下拉列表中选择Cells(细胞)。 |
| 48. | 取消对Use the object name for the file name(使用对象名作为文件名?)的选择。 |
| 49. | 请把文件名设置为cells.cpout。 |
| 50. | 来为“Cytoplasm(细胞质)”重复第47-50步,并设置文件名为cytoplasm.cpout。 |
这些都完成后,ExportToSpreadsheet(导出到电子表格)模块看起来应如图T24.47所示。
注意1:文件后缀".cptoc"代表CellProfiler Table of Contents(CellProfiler内容表格),而".cpout"代表CellProfiler Output(CellProfiler输出)。"Image.cptoc"文件中保存了在管道中处理的所有对象的位置以及图片数目的信息,而"Nuclei.cpout"和"Cells.cpout"分别存储与单个对象以及分析相关的实际列表模式数据。每个在CellProfiler中定义的对象模板都应该有一个独立的".cpout"文件。我们只需要一个"Image.cptoc"文件。
注意2:您.cpout文件的名字必须以您正在导出的对象数据名称开始,可能会只包含对象名称和一般表达式。例如:文件名是nuclei.cpout和nuclei_<regular express>.cpout将产生一个正确的输出,但dataset1nuclei.cpout或者<regularexpression>_nuclei.cpout将导致一个非-工作输出。

Figure T24.47 The second ExportToSpreadsheet Module Set Up to Export nuclei.cpout, cells.cpout, and cytoplasm.cpout.
| 46. | 选择Analyze images(分析图片)来运行管道。你图片所在的文件夹将包括cells.cpout、nuclei.cpout、cytoplasm.cpout以及Image.cptoc文件。该文件夹中,还有对应于平铺数据集中每个原始图片/微孔的一个CellLabel、一个NucleiLabel和一CytoplasmLabel图片。 |
在下一节中,我们将导入和分析平铺或者拼接图片。
