插入带回归分析的散布图
我们将使用前面章节创建的自定义数据表格,来创建一带回归分析的散点图。一旦完成,您将完成数据组(已完成误差线)回归分析的创建,如果门操作进行更改,它也将实时更新。
要开始本教程,您可以使用在“创建自定义数据表格”章节创建的版面。
您也可以打开FCS Express Sample Data(FCS Express样本数据)文件夹中的Titration_Data_Grid_Completed.fey版面,来开始本教程的学习。打开这一版面的所有步骤,均在“创建自定义数据表格”教程中有介绍。
| 1. | 请选中自定义数据网格,选中后它应该有一个绿色边框。 |
| 2. | 请选择Insert(插入)→2D Plots(二维绘图 )→Scatter with regression(带回归分析的散布图)命令(图T17.37)。 |
Figure T17.37 Insert Scatter Plot Command
| 3. | 请在版面上的空白区点击。 |
一个使用自定义数据表格中的滴定数据创建的带回归分析的二维散布图,将出现在版面上,如图T17.38所示。
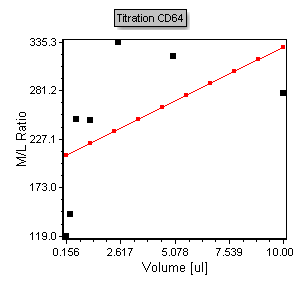
Figure T17.38 Scatter Plot of Titration Data
请注意,该绘图包含黑色的点,这代表这些点来自自定义数据表格。红线数据层是回归分析叠加图层。所有带回归分析的散布图的默认回归拟合,都是线性拟合。在下面的步骤中,我们将调整绘图,让它显示一个更合适的Michaelis-Menten(米氏)回归拟合。我们同样将以调整坐标轴范围来开始,让绘图上的点能更好的拟合。
| 4. | 双击带回归分析的散布图,来调出格式化对话框。 |
| 5. | 请选择Axis(数据轴)项目类别。 |
此时,Formatting scatter plot(格式化散布图)对话框中的Axis(数据轴)页将出现。请参考图T17.39A和图T17.39B来进行以下步骤。
| 6. | 请选择Y Axis(Y轴),如图T17.39A中的蓝色高亮所示。 |
| 7. | 输入“500”到Maximum Range(最大值范围)窗口中,如图中的蓝色高亮所示。 |
| 8. | 请选择X Axis(X 轴),如图T17.39B中的蓝色高亮所示。 |
| 9. | 输入“12”到Maximum Range(最大值范围)窗口中,如图中的蓝色高亮所示。 |
| 10. | 点击Preview(预览)。 |
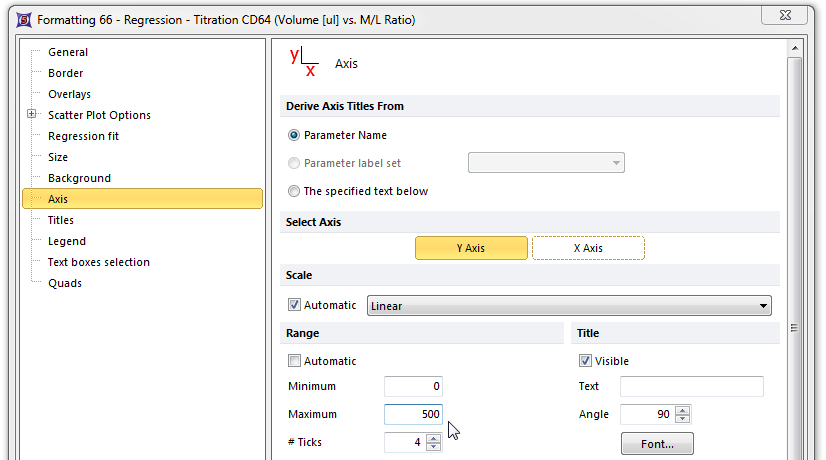
Figure T17.39A Formatting the Y-axis of the Scatter Plot
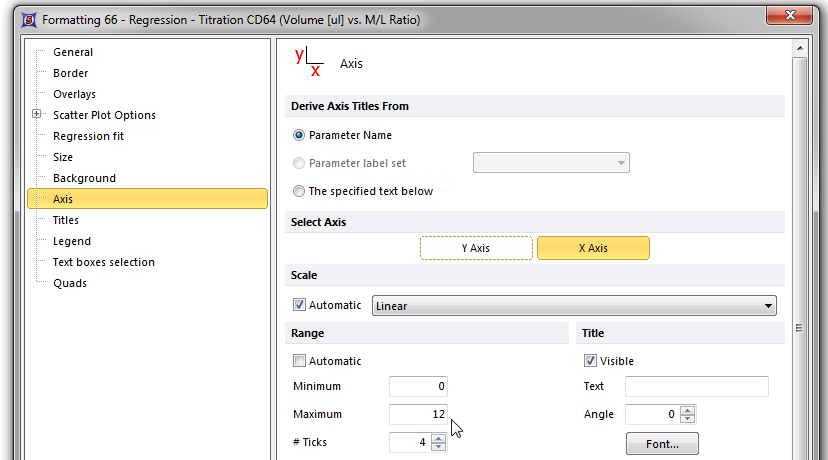
Figure T17.39B Formatting the X-axis of the Scatter Plot
现在,滴定散布图以更合适的坐标系显示于图T17.40中。
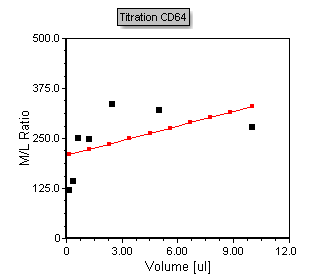
Figure T17.40 Formatted Titration Scatter with regression plot.
现在,我们将更改绘图上的拟合类型,并平滑拟合曲线
| 11. | 请点击Regression fit(回归分析拟合)项目。 |
| 12. | 从Regression Model Options(回归模型选项)→Regression Type(回归分析类型)下拉列表中选择MichMent。 |
| 13. | 将回归分析的点数,从10增加到30。 |
现在绘图看上去应如图T17.41所示。请注意,现在回归曲线拟合相应的数据。
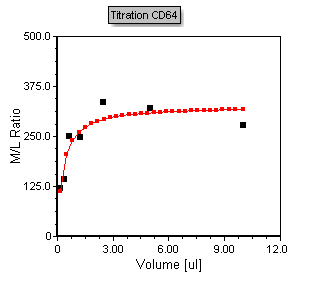
Figure T17.41 - Scatter with regression plot displaying the MichMent regression fit type overlay
现在,我们将增加更过的数据到滴定绘图中,来反映单核细胞群体的变异系数(CV值)。我们将把这些数据作为误差线,输入到散布图上。开始之前,我们将增加一个新的列岛滴定自定义数据表格中,该列是单核细胞CV值数据。
| 14. | 右键点击滴定自定义数据表格中的M/L ratio(M/L比率)列,来调出弹出菜单。 |
| 15. | 请选择Insert(插入)→Column to the Right(把列插入到右侧)命令,如图T17.42中的光标和蓝色高亮所示。 |
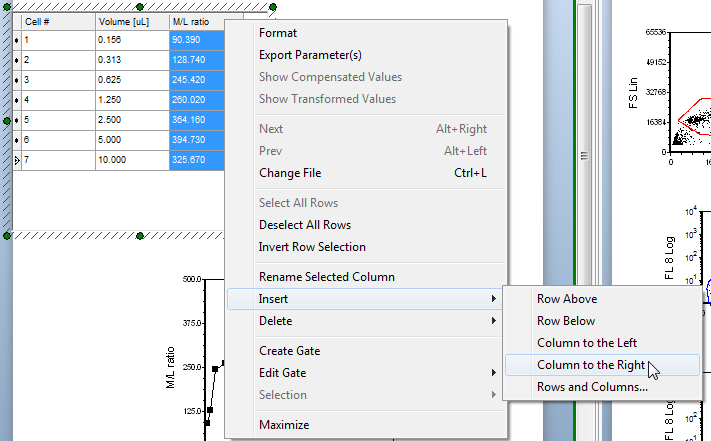
Figure T17.42 Inserting a New Column in the Custom Data Table
此时,New Column Name(新列名)对话框会出现。
| 16. | 输入“Monos CV(单核细胞变异系数)”到新列名字段处。 |
| 17. | 点击OK。 |
此时,一个新的列就会出现在M/L ratio(M/L比率)列的右边,其名称是“Monos CV(单核细胞变异系数)”。现在,我们将插入一代表单核细胞群CD64荧光CV值的标记,到新列中。以下步骤请参考图T17.43。
| 18. | 请点击“Monos CV(单核细胞变异系数)”列最顶上的单元格,让其高亮显示为蓝色。 |
| 19. | 请点击标记图标,如图中的光标所示。 |
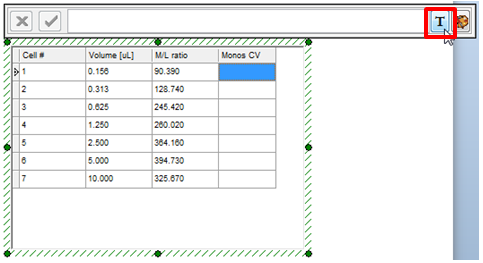
Figure T17.43 Inserting Token Data into a Cell of the New Column
此时,Insert a Token(插入一个标记)对话框将出现。
| 20. | 请选择Statistic(统计)标记。 |
| 21. | 点击Insert(插入)。 |
此时,Create Statistic(创建统计)对话框将出现。
| 22. | 请选择Color Dot - .156ul CD64.fcs.DNS (SS Lin vs.FL 8 Log)。 |
| 23. | 请选择左边的Statistic(统计)项目类别。 |
现在,Create Statistic(创建统计)对话框下的Statistic(统计)页将出现,我们将选择单核细胞门的Y轴CV值作为统计,用于这一标记。以下步骤都针对图T17.44。
| 24. | 请从Statistic:(统计:)列表框中选择Y CV(Y轴变异系数),如图中的蓝色高亮所示。 |
| 25. | 请从Gate:(门:)列表框中选择Monos(单核细胞门),如图中的光标和蓝色高亮所示。 |
| 26. | 点击OK。 |
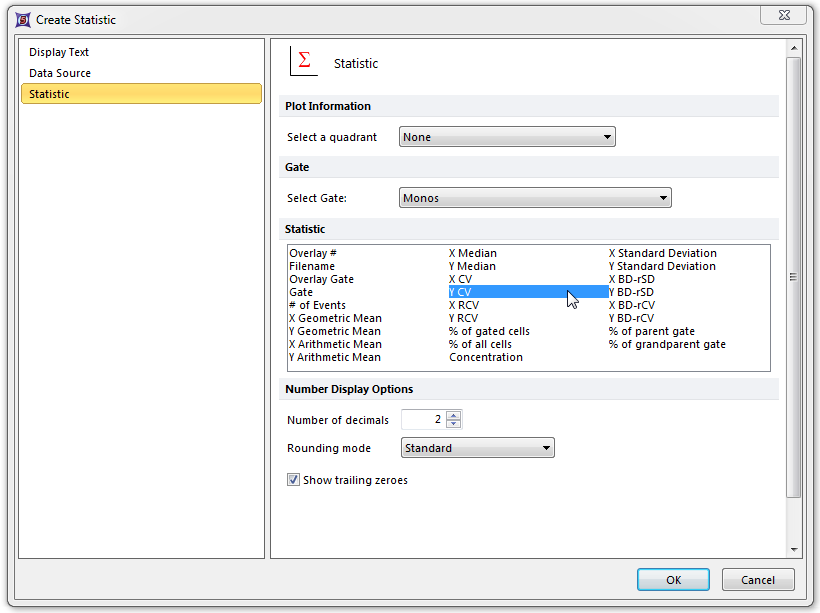
Figure T17.44 Create Statistic Dialog for Monos CV
现在,滴定自定义数据表格进行了更新,来反映0.156μl数据的“Monos CV(单核细胞变异系数)”统计标记,如图T17.45中的蓝色高亮所示。
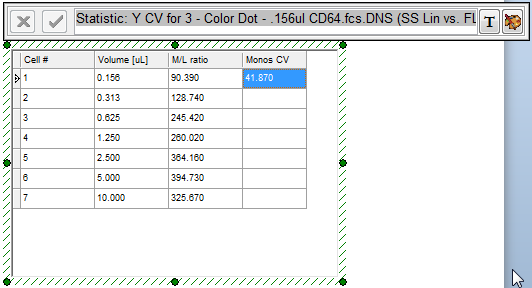
Figure T17.45 Monos CV Token Data
用其他CD64体积的数据,来继续填充“Monos CV(单核细胞变异系数)”列,重复地第18-26步。或者,您可以使用组合的复制和粘贴命令,如前面章节演示的一样,利用合适的数据绘图,来确保标记的更新。当“Monos CV(单核细胞变异系数)”列填充满后,看上去应如图T17.46所示。
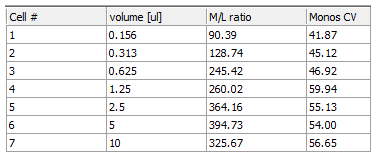
Figure T17.46 Titration Custom Data Table with Monos CV Column Completed
在滴定散布图中,我们将使用单核细胞变异系数作为误差线。
| 27. | 双击带回归分析的散布图,来调出格式化对话框。 |
此时,Formatting Overlays(格式化数据层)对话框会出现。
| 28. | 请选择Overlays(数据层)项目类别。 |
| 29. | 从Error Bar Param(误差线参数)下拉列表中选择“Monos CV(单核细胞变异系数)”(图T17.47)。 |
| 30. | 点击OK。 |
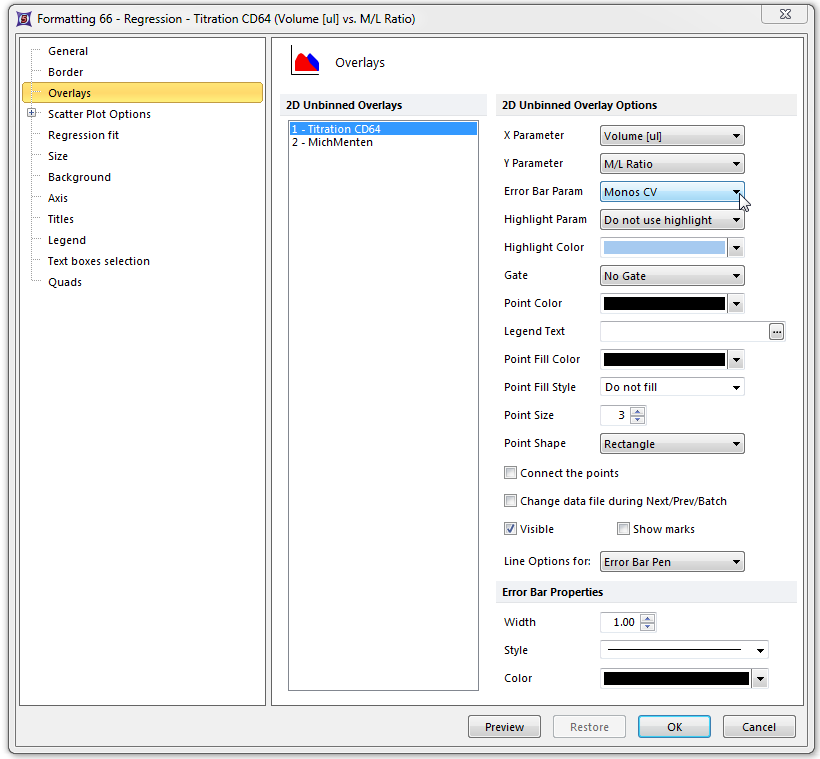
Figure T17.47 Formatting Overlays Dialog for the Titration Scatter Plot
现在,滴定散布图进行了更新,来反映添加的蓝色误差线(使用单核细胞变异系数数据),如图T17.48所示。请注意,当您调整版面上的任何门时,散布图上的数据点、回归分析和误差线都将实时更新。
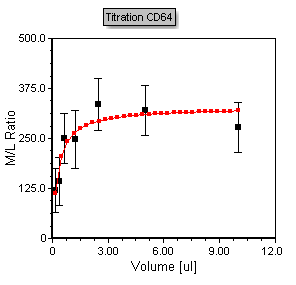
Figure T17.48 Titration Scatter Plot with Error Bars
