
导出单个图片 |

|

|

|
|
|
导出单个图片 |

|

|

|
导出单个图片
|
|
||
在以下两节中,你将学习:
| • | 如何从CellProfiler中导出不是从96孔板中获取的一组单个图片。 |
| • | 如何把数据导入到FCS Express第4版中。 |
这一部分适用于单个图片数据组,这组图片可能不是从96孔板上采集的,而是从一个玻片上拍下来的。
在这一节中我们要用到的向FCS Express中导出数据的CellProfiler模块包括: LoadImages(导入图片)模块、ConvertObjectsTo Images(把对象转化为图片模块)、SaveImages(保存图片)模块以及ExportToSpreadsheet(导出到电子表格)模块。本例中最后完成的管道文件可以在教程样本数据库中找到,其名称是Section2pipelineCOMPLETED.cp。这个完整的、作为模板的管道可以用来和用户的管道进行比较。
我们要使用的样本数据集中的图片可在 教程样本数据库中找到,其名称是"5 and 10 minute time point images(5分钟和10分钟时间点的图片)"。这些图片是基于CellProfiler样本中的样本实验"Human cytoplasm-nucleus translocation assay (SBS Bioimage) (人类细胞质-核易位实验(SBS Bioimage))" 。

我们将把从CellProfiler中导出数据的步骤依据模块划分如下。为了向FCS Express中导出做准备,导入Section2pipeline.cp后,请遵循以下步骤来修改管道。
选择默认的输入和输出文件夹
要让FCS Express正确地组织数据,首先请保证在CellProfiler中默认的输入文件夹(存储图片的地方)以及输出文件夹是同一个文件夹(在Input/Output Folder(输入/输出文件夹)窗口中进行设置)(图T24.13)。
注意: DefaultOUT.mat文件也将被导出到Output Folder(输出文件夹)中。这个文件可在MATLAB中使用。如果不想用这个文件,我们建议将其删除,以节省硬盘空间。

图T24.13 Set the Default Input and Output Folders to the Same Location (把输入和输出文件夹设置为同一个文件夹)
设置LoadImages Module(导入图片模块)
要想让FCS Express通过管道读出数据,图片文件名中的元数据必须被提取出来。我们必须定义一个Regular Expression(一般表达式),用以在下面步骤中要使用到的文件名或数据路径中找到元数据(图T24.14):
| 1. | 请从Extract metadata from where?(从哪里提取元数据?)下拉列表中点击Channel2-。 |
| 2. | 请选择你想要使用的元数据。对本例来说,请选择File name(文件名称)。 |
| 3. | 请在Regular expression that finds metadata in the file name.(在文件名中寻找元数据的一般表达式。)字段中输入你想要使用的"Regular Expression(一般表达式)"。 在本例中,我们将使用"Channel2-[0-9]{2}-(?P<Row>[A-H])-(?P<Column>[0-9]{2}).tif",该表达式根据图片名中的行字母以及列数字定义了图片在细胞板上的位置。 |
注:
| • | 我们只需为数据集中的一个图片设置元数据以及一般表达式。在本例中,我们为第2个通道做了这些设置。 |
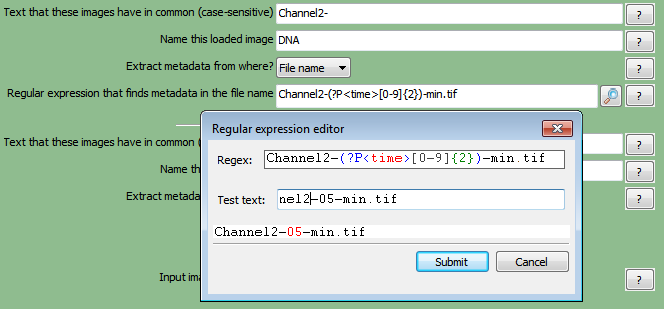
图T24.14 Setting Up a Regular Expression in the LoadImages Module (在导入图片模块中设置一般表达式)
ConvertObjectsToImage Modules (把对象转化为图片模块)
在该模块中,模块"Identify Primary/Secondary Objects(确认主要/次要对象)"中定义好的对象将被转化为FCS Express在导入和分析中可以使用的图片蒙版。图T24.15给出了一个该模块窗口的例子。
| 4. | 请选择管道中的第一个ConvertObjectsToImage(把对象转化为图片)模块 。 |
| 5. | 请从Select the input objects(选择输入对象)下拉列表中选择Nuclei(细胞核)。 |
| 6. | 请在Name the output image(为输出图片命名)字段中选择一个名字。例如,可以使用"Nuclei(细胞核)"。 |
| 7. | 请在Select the color type(选择颜色类型)下拉菜单中选择颜色类型,请选择uint16(图T24.15)。 |
| 8. | 请在管道中选择第二个ConvertObjectsToImage(把对象转化为图片)。 |
| 9. | 请在Select the input object(选择输入对象)下拉列表中选择Cells(细胞)。 |
| 10. | 请在Name the output image(为输出图片命名)字段输入想要的名称。本例中,我们将使用"Cells(细胞)"。 |
| 11. | 请在Select the color type(选择颜色类型)下拉菜单上选择颜色类型,请选择uint16。 |
注:在定义你自己的管道时,请为通过Identify Primary/Secondary Objects(确认主要/次要对象)模块定义的对象重复第4步到第7步。
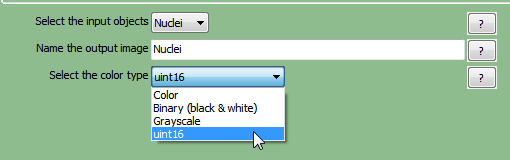
图T24.15 ConvertObjectsToImage Module: Defining Nuclei and Cells Image Masks (把对象转化为图片模块: 定义细胞核以及细胞图像蒙版)
SaveImages Module (保存图片模块)
我们在ConvertObjectsToImage(把对象转化为图片)模块中定义的图像蒙版必须被保存下来并且和原始的图像/数据进行关联。
| 12. | 请选择管道中的第一个SaveImages(保存图片)模块。 |
| 13. | 请在Select the type of image to save(选择保存的图片类型)下拉列表中选择Image(图像)。 |
| 14. | 请在Select the image to save(选择要保存的图片)中选择Cells(细胞)。 |
注: 定义自己的管道时,请选择在ConvertObjectsToImage(把对象转化为图片)模块中定义的相应的图片。
| 15. | 请在Select method for constructing file names(创建构建文件名的方法)下拉列表中选择Single name(单个文件名)。 |
| 16. | 请在Enter single file name(输入单文件名)字段输入任何你想要的名字,然后再输入一个连字符"-"。在本例中,我们使用CellLabel-。 |
| 17. | 请在CellLabel-后右键点击,然后选择Time(时间)。 |
| 18. | 请选择管道中的第二个SaveImages(保存图片)模块。 |
| 19. | 请从Select the type of image to save(选择保存的图片类型)下拉菜单中选择Image(图片)。 |
| 20. | 请从Select the image to save(选择要保存的图片)中选择Nuclei(细胞核)。 |
| 21. | 请在Select method for constructing file names(创建构建文件名的方法)下拉列表中选择Single name(单个文件名)。 |
| 22. | 请在Enter single file name(输入单个文件名)字段输入任何你想要的名字,然后再输入一个连字符"-"。在本例中,我们将使用NucleiLabel-。 |
| 23. | 请在NucleiLabel-后右键点击,选择time(时间)。 |
注: 这是为导入图片模块把"时间"值定义为一般表达式。使用一般表达式将为每个输出文件根据输入文件名的文本设置一个独一无二的名字。这将防止输出文件间的相互覆盖, 并让FCS Express可以找到每个图片文件对应的图片蒙版。
请为CellLabel以及NucleiLabel的SaveImages(保存图片)模块执行以下的第24到第30步。
| 24. | 请从Select the format to use(选择使用的格式)下拉列表中选择tif。 |
| 25. | 请在Image bit depth(图片比特深度)下拉列表中选择16。 |
| 26. | 请从Output file location(输出文件位置)选择Default Output Folder(默认输出文件夹)。 |
| 27. | 请点选Overwrite existing files without warning?(覆盖已有文件时不进行警告?)复选框。 |
| 28. | 请从Select how often to save(选择如何保存)下拉列表中选择Every cycle(每个周期)。 |
| 29. | 请从Select colormap(选择颜色图)下拉列表中选择gray(灰度)。 |
| 30. | 请勾选Update file names within CellProfiler?(在CellProfiler里更新文件名?)复选框。 |
这时CellLabel(细胞标记)的SaveImages(保存图片)模块看起来应该如图T24.16所示。
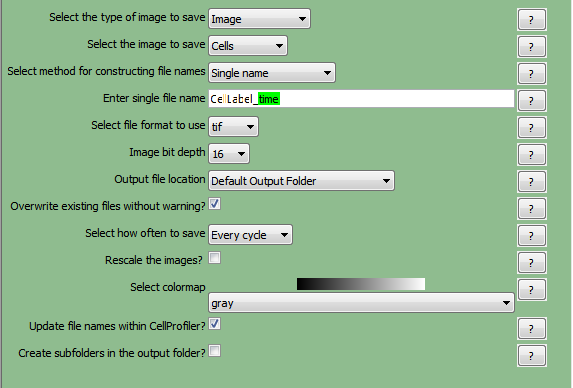
图T24.16 Defining the SaveImages Module (定义保存图片模块)
ExportToSpreadsheet Module (导出到电子表格模块)
既然所有的图片以及对象图片模板都已经定义并保存好了,用户现在即可让CellProfiler导出用户想要在FCS Express中查看的测量数据。在本例中,我们将导出所有的测量数据。
| 31. | 请选择ExportToSpreadsheet(导出到电子表格)模块。 |
| 32. | 请从下拉列表中选择Comma(逗号)(",")作为列分隔符。 |
| 33. | 请取消对Prepend the output file name to the data file names?(在数据文件名称前追加数据文件名?)复选框的勾选。 |
| 34. | 请从Output file location(输出文件位置)下拉列表中选择Default Output Folder(默认输出文件夹)。 |
| 35. | 请取消对Export all measurements?(导出所有测量数据?)的点选。 |
| 36. | 请从第一个Data to export(要导出的数据)下拉列表中选择Image(图片)。 |
| 37. | 请取消对Use the object name for the file name?(为文件名使用对象名?)的勾选。 |
| 38. | 请把文件名设置为Image.cptoc。 |
| 39. | 请选择Add another data set(添加另外一组数据集)。 |
| 40. | 请从第二个Data to export(要导出的数据)下拉列表中选择Nuclei(细胞核)。 |
| 41. | 请取消对Use the object name for the file name(使用对象名作为文件名)的勾选。 |
| 42. | 请把文件名设置为nuclei.cpout。 |
| 43. | 请为"Cells(细胞)"重复第39-42步,把文件名设置为cells.cpout。 |
这些都做完后,ExportToSpreadsheet(导出到电子表格)模块应该看起来如图T24.17所示。
注意: 文件后缀".cptoc"代表CellProfiler Table of Contents(CellProfiler内容表格), 而".cpout"代表CellProfiler Output(CellProfiler输出)。"Image.cptoc"文件中保存着在管道中处理的所有对象的位置以及图片数目的信息, 而"Nuclei.cpout"和"Cells.cpout"分别存储和单个对象以及分析相关的实际列表模式数据。每个在CellProfiler中定义的对象模板都应该有一个独立的".cpout"文件。我们只需要一个"Image.cptoc"文件。

图T24.17 ExportToSpreadsheet Module (导出到电子表格模块)
| 44. | 请选择Analyze images(分析图片)来运行管道。图片所在的文件夹将包括每个时间点对应的cells.cpout、nuclei.cpout以及Image.cptoc文件。该文件夹中还有对应于实验中每个原始图片的一个CellLabel以及一个NucleiLabel图片(图T24.18)。 |

图T24.18 Example of Output Folder once CellProfiler Analysis is Complete (CellProfiler分析完成后的输出文件夹实例)
在下一节中,我们将把单个图片实验导入到FCS Express中。