
对进行分裂的T-细胞进行拟合 |

|

|

|
|
|
对进行分裂的T-细胞进行拟合 |

|

|

|
对进行分裂的T-细胞进行拟合
|
|
||
在本节中,我们将:
| • | 学习如何把曲线拟合算法应用于柱状图。 |
| 1. | 请选中Insert(插入)→1D Plots(一维绘图)→Proliferation(细胞分裂)命令(图T20.12)。 |
图T20.12 Inserting a Proliferation Plot (插入细胞分裂绘图)
| 2. | 请在版面上想要添加柱状图的地方点击。 |
如果出现Select a Data File(选择数据文件)对话框,请选择001-T only CFSE+CD3PE+SB.fcs文件。或者, 因为这个文件是最近使用的文件,新绘图应该自动显示这个文件上的数据。
| 3. | 请为水平数轴选中FITC-A来显示CFSE染色。 |
| 4. | 请从下拉列表中选择Gating(门操作)→Create Gates(创建门)→Current Gate(当前门)→Final T(Final T门)来在柱状图上应用Final T门。 |
如果绘图是在线性坐标系上,请把它改为对数坐标,方法是选择Format(格式)→Plot Options(绘图选项)→Axes(数轴)命令,然后在Formatting Axes(格式化数轴)对话框中的下拉列表中选择Log(对数)。
最初的对柱状图的曲线拟合应该看起来如图T20.13所示。这是对未进行分裂的对照细胞的曲线拟合结果。
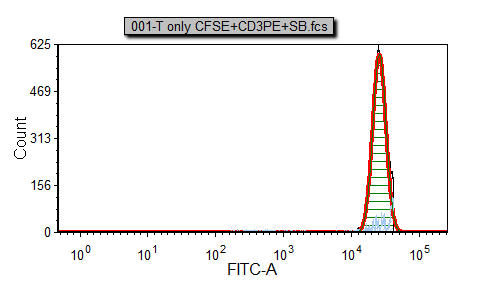
图T20.13 Proliferation Fitting to Control Histogram (对"对照柱状图"进行细胞分裂拟合)
现在让我们来导入一个有分裂细胞的文件。
| 5. | 请选择Data(数据)→Change Data on Selection(修改选中的数据)→Next(下一个)命令向前翻一个文件到002- T+DC 4to1 CSFE+CD3PE+SB.fcs文件。 |
这时的曲线拟合柱状图看起来应该如图T20.14所示。
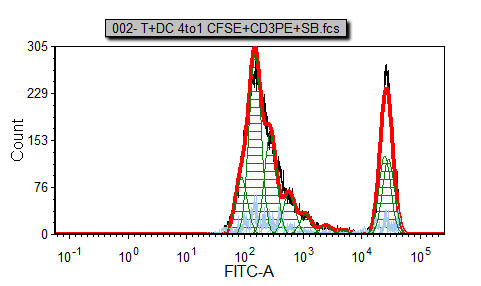
图T20.14 Initial Curve Fitting on Histogram of Proliferating Cells (对正在分裂的细胞进行拟合的初始曲线)
细胞分裂曲线拟合的默认颜色如下:
黑色 – 柱状图整体轮廓。它包括所有数据而且不代表任何拟合算法的输出。
红色 – 被拟合的数据的整体轮廓。该轮廓排除了算法认为是噪声的那部分信号。
浅蓝色 – 噪声。这是算法判定为噪声信号的那部分,这部分信号不包括在统计计算中。
绿色 – 模型拟合的分裂细胞群。这是对数据拟合后的实际曲线,代表父代细胞、未分裂细胞群和所有正在进行分裂的细胞代。
注意到在图T20.14中有显示为浅蓝色的细胞,它们被模型认为是噪声信号。理想情况下,越少细胞被认为是噪声信号越好。现在我们要来修改一下细胞分裂拟合参数,尝试能否改进对模型的拟合。
| 6. | 请右键点击柱状图,调出相关弹出菜单。 |
| 7. | 请从弹出菜单上选择Format(格式)。 |
这时会出现Formatting(格式化)对话框(图T20.15)。
| 8. | 请从对话框左边选择Proliferation(细胞分裂)→Fit Parameters(拟合参数) |
| 9. | 请不要选择Same CV for All Peaks(所有信号峰使用相同的变差系数) |
| 10. | 请点击OK。 |

图T20.15 Formatting Dialog -- Proliferation Fit Parameters Category (格式化对话框 -- 细胞分裂拟合参数类别选项)
这时细胞分裂柱状图应该看起来如图T20.16所示。
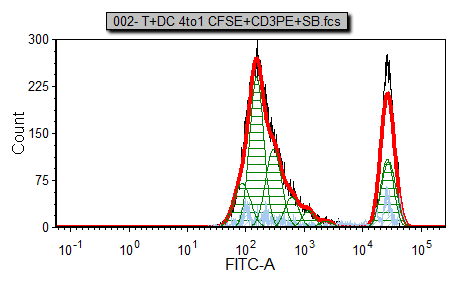
图T20.16 Proliferation Histogram with Improved Proliferation Fit (细胞分裂柱状图上改进过的细胞分裂拟合)
现在的拟合比以前要好一些了。注意到红色的曲线(拟合轮廓线)和黑色的曲线(原始数据轮廓线)靠得更近了。但是为了更好地考察拟合的好坏,我们需要使用统计数据,也就是我们现在要讨论的内容。
技术介绍: 以下技术注释将简短地描述拟合过程是如何实现的。这些信息可以更好地帮助用户理解如何调整影响拟合的参数。如果不想了解这些细节,请直接跳过。
|
| 11. | 请选中Data(数据)→Change Data on Selection(修改选中的数据)→Next(下一个)命令,向下翻一个文件来到文件003- T+DC 64to1 CSFE+CD3PE+SB.fcs。 |
这时对柱状图的细胞分裂拟合应该看起来如图T20.17所示。注意,和前个文件不同,在这个样本中只有很少的细胞已经进行了分裂。
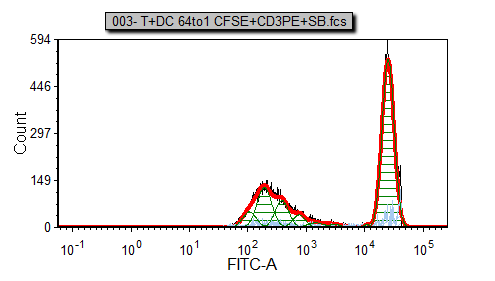
图T20.17 Proliferation Fit Model on Sample 003- T+DC 64to1 CSFE+CD3PE+SB.fcs (针对003- T+DC 64to1 CSFE+CD3PE+SB.fcs文件的细胞分裂拟合模型)
在某些情况下,用户想要使用对照柱状图中的未分裂的细胞峰的峰值。尽管在本例中我们不需要这个值,我们还是来学习一下这个功能。
| 12. | 请选择Insert(插入)→1D Plots(一维绘图)→Histogram(柱状图)命令来插入一个新的柱状图(而非曲线拟合柱状图)。 |
| 13. | 请在版面上想要添加柱状图的地方点击。 |
如果柱状图自动导入当前数据文件:
| 14. | 请选择Data(数据)→Change Data on Selection(修改选中的数据)→Previous(前一个)命令。 |
| 15. | 请点击Previous(前一个)按钮两次,回退两个文件到对照文件001-T only CFSE+CD3PE+SB.fcs。 |
| 16. | 请把Final T门应用到柱状图上。 |
| 17. | 请把x-坐标参数修改为FITC-A。 |
如果柱状图没有自动导入并出现了Select a Data File(选择数据文件)对话框,那么:
| • | 请从Select a Data File(选择数据文件)对话框选中001-T only CFSE+CD3PE+SB.fcs。 |
| • | 请从Open Histogram(打开柱状图)对话框中的参数列表上选择FITC-A。 |
| • | 请从Open Histogram(打开柱状图)对话框上的下拉门列表中选中Final T。 |
| 18. | 请把柱状图的x-轴坐标设置为Log(对数)。 |
这时的对照柱状图看起来应该如图T20.18所示。
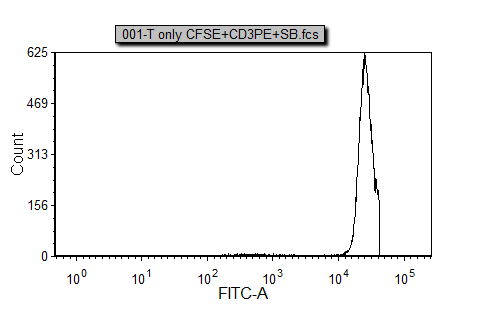
图T20.18 Control Histogram (对照柱状图)
| 19. | 请右键点击文件003- T+DC 64to1 CSFE+CD3PE+SB.fcs对应的细胞分裂柱状图,调出相关的弹出菜单。 |
| 20. | 请从弹出菜单上选择Format(格式)。 |
| 21. | 请在对话框的左边选中Proliferation(细胞分裂)→Fit Parameters(拟合参数) |
| 22. | 请选择Use Fixed Starting Generation(使用固定的起始细胞代) |
| 23. | 请删除Use Fixed Starting Generation(使用固定的起始细胞代)字段中的内容。 |
| 24. | 请点击Use Fixed Starting Generation(使用固定的起始细胞代)旁边的Token(标记) |
这时会出现Insert a Token(插入标记)对话框(图T20.19)。
| 25. | 请在列表顶端选择Statistic(统计),如图T20.19中的光标所示。 |
| 26. | 请点击Insert(插入)。 |
.png)
图T20.19 Insert a Token Dialog (插入标记对话框)
这时会出现Create Statistic(创建统计)对话框,Data Source(数据源)选项被选中(图T20.20)。
| 27. | 请选中对照柱状图,如图T20.20中的蓝色高亮所示。注意到所选柱状图的预览出现在右方。 |
| 28. | 请从左边的列表中选择Statistic(统计)列表选项(图T20.20)。 |
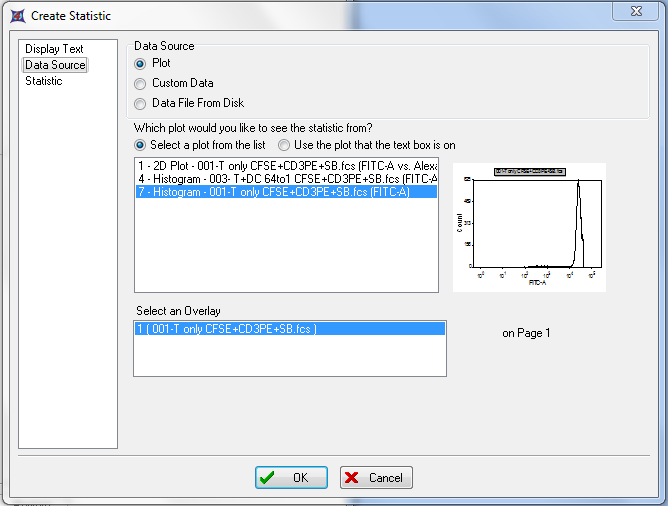
图T20.20 Create Statistic Dialog -- Data Source (创建统计对话框 -- 数据源)
这时会出现Create Statistic(创建统计)对话框中的Statistic(统计)页面(图T20.21)。
| 29. | 请从Statistic(统计)选项中选择Median(中间值),如图T20.21中的蓝色高亮所示。 |
因为柱状图已经应用了门,我们无需再指定门。
| 30. | 请点击OK。 |
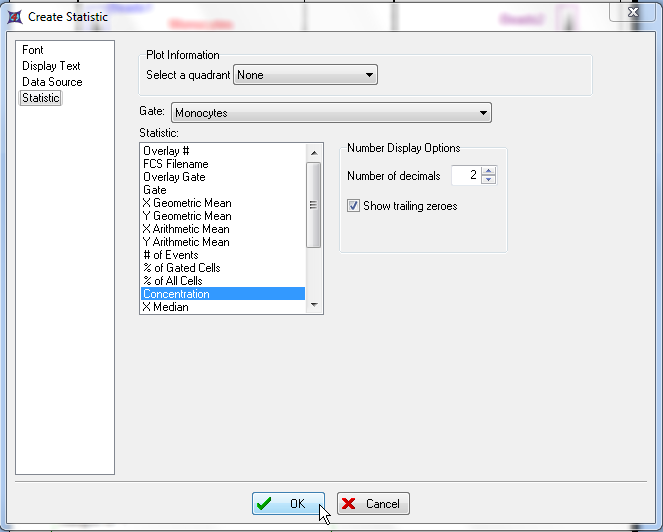
图T20.21 Create Statistic Dialog -- Statistic Category (创建统计对话框 -- 统计类别选项)
这时的Formatting(格式化)对话框将会显示Use Fixed Starting Generation(使用固定的起始细胞代)字段中含有我们刚刚创建的标记统计(柱状图的中间值)。
| 31. | 请点击OK键来关闭Formatting(格式化)对话框。 |
这时的细胞分裂柱状图003- T+DC 64to1 CSFE+CD3PE+SB.fcs将进行更新,显示曲线拟合算法已经使用了对照柱状图的中间值作为起始细胞代位置,而不是让曲线拟合算法计算起始细胞代细胞群的位置。
实际上拟合并未改变多少。这是因为曲线拟合算法正确地找到了细胞分裂样本中的起始细胞代。但在某些情况下,手动设置对照样本中的起始细胞代还是很重要的。而且有时在CFSE实验中,对照样本的信号峰的荧光值会和实验样本中的未分裂细胞的信号峰的荧光值有所偏差。这些偏差有时就已经可以让用户必须放弃使用对照柱状图来设置未分裂细胞的位置了。如在上面的技术介绍中提到的那样,选择正确的设置在取得可靠的拟合过程中非常关键。
接下来,我们将对细胞分裂柱状图进行格式化以及显示统计。