
定义目标细胞群 |

|

|

|
|
|
定义目标细胞群 |

|

|

|
定义目标细胞群
|
|
||
在本节中,我们将:
| • | 学习如何在T-细胞群上应用门。 |
| 1. | 请打开一个新的空白版面。 |
| 2. | 请从FCS Express Sample Data(FCS Express样本数据)文件夹中插入001-T only CFSE+CD3PE+SB.fcs对应的PE-A与SSC-H之间的点状图。要更好地了解如何打开一个绘图,请查看插入绘图教程。 |
数轴的默认设置是线性坐标。但因为这组数据已经经过补偿,查看数据的最好方法是用一个在数值较低区是线性、数值较高区是对数的坐标系。在此我们将为x-轴使用HyperLog(超对数)设置。要更多的了解FCS Express支持的各种坐标系,请参考坐标系一节。
| 3. | 请选中Format(格式)→Plot Options(绘图选项)→Axes(坐标轴)命令。 |
这时会出现Formatting Axes(格式化坐标轴)对话框,如图T20.1所示。
| 4. | 请选中X-axis(X-轴),如图T20.1中的蓝色高亮所示。 |
| 5. | 请从Scale(坐标系)下拉列表中选中HyperLog(超对数坐标系),如图T20.1中的光标所示。 |
| 6. | 请点击OK。 |
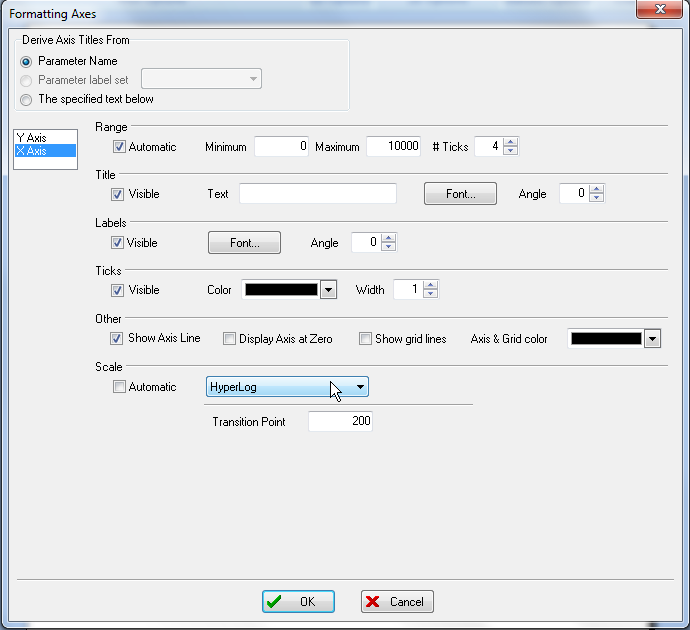
图T20.1 Formatting Axes Dialog (格式化数轴对话框)
现在点状图应该已经进行了更新,显示x-轴的超对数坐标。样本中的染色剂是CD3-PE,因此我们将在CD3-阳性细胞上应用一个门来定义T-细胞群,并排除那些有很大侧向分散信号的细胞。
| 7. | 请创建一个包围CD3+的T-细胞的多边形门(图T20.2)。 |
| 8. | 请把这个门命名为"CD3+T"。 |
这个门看起来应该和Figure T20.2中显示的门相似(要想更多地了解如何创建门,请参考门操作教程)。

图T20.2 T-cell Gate on CD3+ Cells (应用在CD3+细胞上的T-细胞门)
样本中也使用了染色剂Sytox Blue,该染色剂和碘化丙啶类似,可以帮助我们排除那些被Sytox Blue染色的"死"细胞。
| 9. | 请点击纵向坐标轴名"SSC-H",把它改为代表Sytox Blue染色的"AlexaFluor 405-A"。 |
| 10. | 请把y-轴坐标改为HyperLog(超对数)。请参考前边的第3步到第6步,只是在此要使用y-轴,而非x-轴。 |
我们现在要在CD3PE与Sytox Blue的绘图上应用以前创建的CD3+T门来初步定义T-细胞,如图T20.2所示。
| 11. | 如图点状图并非处于选中状态,请点击来将其选中。 |
| 12. | 请从下拉列表中选中Gating(门操作)→Create Gates(创建门)→Current Gate(当前门)→CD3+T(图T20.3)。 |

图T20.3 Selecting the CD3+T Gate (选中CD3+T门)
| 13. | 请从下拉列表中选中Format(格式)→Change(修改)→Change Plot Type(修改绘图类型)→Density(密度图)(图T20.4)。 |
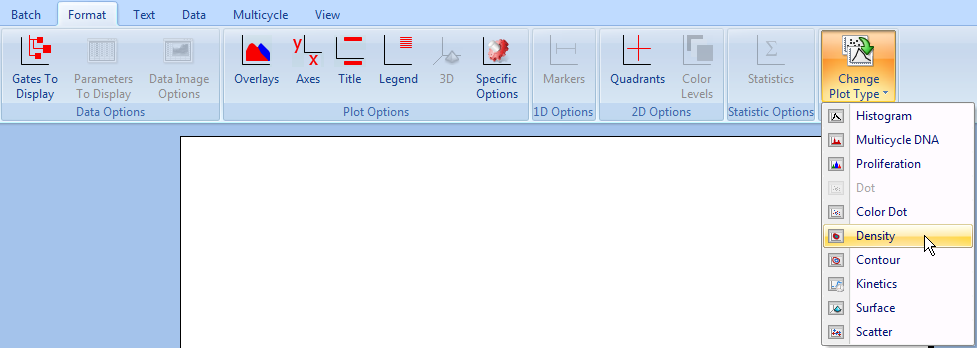
图T20.4 Changing the Plot Type to Density (把绘图类型修改为密度图)
这时的散点图显现为密度图(图T20.7A)。我们现在要来修改密度图的分辨率。
| 14. | 请选中Format(格式)→Plot Options(绘图选项)→Specific Options(特定选项)命令(图T20.5)。 |
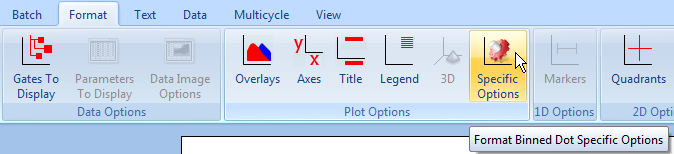
图T20.5 Selecting Format Plot Specific Options (选中格式化绘图特定选项)
这时会出现Formatting Binned Plot Specific Options(格式化区间绘图特定选项)对话框(图T20.6)。
| 15. | 请从对话框中的Resolution Options(分辨率选项)页上的Resolution(分辨率)下拉列表中选择512 x 512,如图T20.6中的光标所示。 |
| 16. | 请点击OK。 |
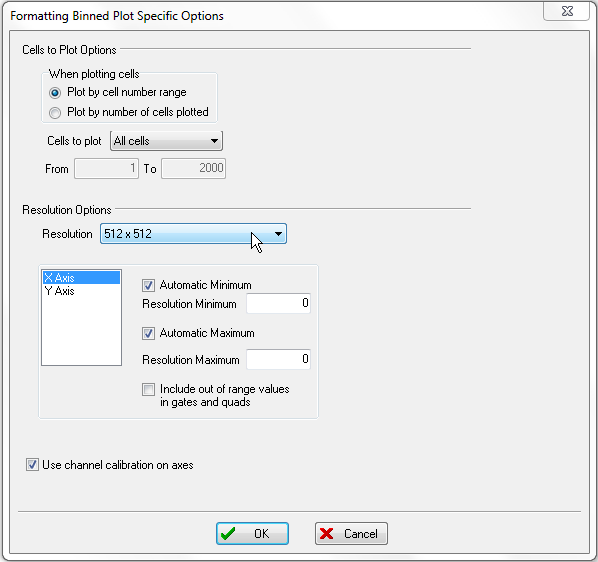
图T20.6 Formatting Binned Plot Specific Options Dialog (格式化区间化绘图特定选项)
这时会出现这个密度图,显示512 x 512分辨率,如图T20.7B所示。
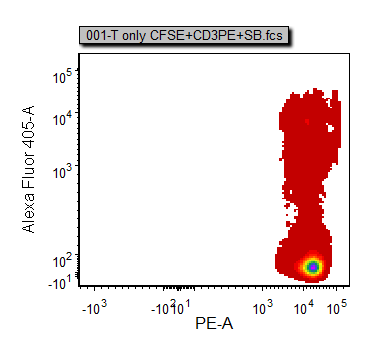
图T20.7A Density Plot with Default Resolution (密度图,默认分辨率)
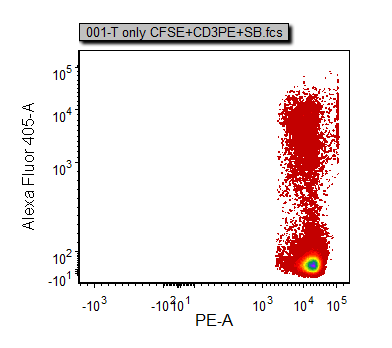
图T20.7B Density Plot with 512 x 512 Resolution (密度图,分辨率为512 x 512)
| 17. | 如果密度图并非处于选中状态,请点击来将其选中。 |
| 18. | 请选中Data(数据)→Change Data on Selection(修改选中的数据)→Next(下一个)命令。 |
| 19. | 请再次点击Next(下一个)按钮,向前翻两个文件,来到003- T+DC 64to1 CFSE+CD3PE+SB.fcs文件。 |
这时密度图会进行更新来显示新的数据文件。我们将使用这些数据来进一步定义使用Sytox Blue的T-细胞门,以排除"死"细胞。
| 20. | 请在CD3阳性以及Sytox Blue阴性细胞周围创建一个矩形门如图T20.8所示。 |
| 21. | 请把门命名为CD3+SB-。 |
这时绘图和门看起来应该如图T20.8所示。

图T20.8 Density Plot with CD3+SB- Gate to Further Define T-cells (在密度图上创建CD3+SB-门来进一步定义T-细胞)
现在我们要为CD3+SB-门创建一个子门CD3+T。通过在Sytox Blue阴性细胞上创建门,我们已经排除了死细胞,因为Sytox Blue染色剂只可以进入细胞膜缺乏抵抗力的细胞(这些细胞被认为是死细胞)。
| 22. | 请选中Insert(插入)→General(常规)→Gate View(门视图)命令。 |
| 23. | 请在版面上你希望Gate View(门视图)窗口出现的地方点击。 |
这时门视图窗口会出现在版面上(图T20.9A)。
| 24. | 请拖到CD3+SB-门,将其作为CD3+T的子门。 |
| 25. | 请点击CD3+T门旁边的"+"图标,展开列表。 |
这时CD3+SB-门已经是CD3+T门的子门了,门视图窗口将看起来如图T20.9B所示。
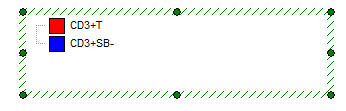
图T20.9A Initial Gate View Window (开始时的门视图窗口)

图T20.9B Gate View with CD3+SB- as a Child Gate (门视图中CD3+SB-作为子门)
如果密度图并非处于选中状态,请点击来将其选中。
| 26. | 请从下拉列表中选中Gating(门操作)→Create Gates(创建门)→Current Gate(当前门)→CD3+SB-。 |
这时的密度图将进行更新,显示CD3+SB-门。
| 27. | 请选中Data(数据)→Change Data on Selection(修改选中的数据)→Previous(前一个(项))命令。 |
| 28. | 请点击Previous(前一个(项))按钮两次,向后移动两个文件到001-T only CFSE+CD3PE+SB.fcs文件。 |
这时绘图应该回到了001-T only CFSE+CD3PE+SB.fcs。
| 29. | 请把密度图的水平坐标轴改为FITC-A。这个坐标代表CFSE染色。 |
| 30. | 请创建一个门来排除较高的CFSE (FITC-A)值但却包括所有其它事件,如图T20.10所示。 |
在这个例子中的有着比主要细胞群较高CFSE值的细胞是非自然信号,应该被排除。
| 31. | 请把这个门命名为"Final T"。 |
这时的绘图和门看起来应该如图T20.10所示。
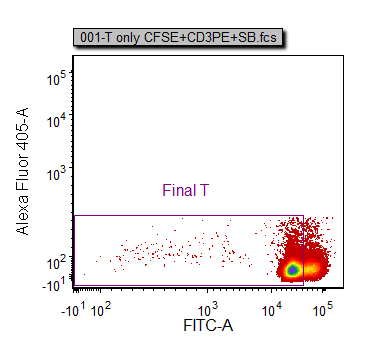
图T20.10 Density Plot of CFSE vs Sytox Blue with Final T Gate Defined (CFSE以及Sytox Blue密度图以及绘图上定义的最后的T细胞门)
这时门视图也应该进行更新来显示Final T门。
| 32. | 请把Final T门拖为CD3+SB-门的子门。 |
| 33. | 请点击CD3+SB-门旁边的"+"号来展开列表。 |
最后的Gate View(门视图)窗口应该看起来如图T20.11所示。

图T20.11 Gate View with Final Gate Relationshps (门视图上最后的各个门之间的关系)
注意到在图T20.10中,Final T门中的事件显示出CFSE荧光强度有一个范围。这些是正在分裂的T-细胞。在下一节中,我们将学习如何对正在分裂的T-细胞进行拟合。