拟合增殖细胞
在本节中,我们将:
| • | 学习如何把曲线拟合算法和参数应用于柱状图。 |
| • | 使用标记来定义一Fixed Starting Generation(固定起始细胞代数)和Background(背景)。 |
| • | 插入增殖位置标识。 |
我们将通过导航这些数据来开始,从而知道这实验看上去像什么和覆盖的颜色编码代表什么。
| 1. | 打开教程样本数据文件夹下"Proliferation Tutorial(增殖教程)"文件夹中的"Proliferation Tutorial Fitting Proliferating Cells Section.fey"版面。 |
| 2. | 导航到Proliferation Data(增殖数据)页面标签。 |
您会注意到,在Proliferation Data(增殖数据)页面标签上有一针对10 uM CellTrace Violet Stimulated Control.fcs样本的增殖绘图(图T20.5a)。这是针对阳性对照-增殖细胞的曲线拟合结果。在数据列表中,有4个样本。
我们现在将切换到没有增殖的对照样本处。
| 3. | 请选择Data(数据)标签→ Change Data on All(更改所有数据)→ Previous(前一项)命令,来导入数据列表中的前一个文件,即10 uM CellTrace Violet Control.fcs文件(图T20.5b)。 |
注意:10 uM CellTrace Violet Control.fcs文件代表对为分裂的细胞进行染色的对照样本,可以作为增殖峰的开始。
我们现在将切换到刺激后的样本上。
| 4. | 请选择Data(数据)标签→ Change Data on All(更改所以数据)→ Next(下一项)命名,来前进两个文件到10 uM CellTrace Violet Stimulated Sample.fcs文件(图T20.5c)。 |
注意:10 uM CellTrace Violet Stimulated Sample.fcs文件代表我们刺激后(正在分裂)的样本,我们将测量这些样本的增殖程度。
我们现在将切换到未染色的对照样本上。
| 5. | 请选择Data(数据)标签→ Change Data on All(更改所以数据)→ Next(下一项)命令,来到数据列表最下面的Unstained Control.fcs文件(图T20.5d)。 |
注意:Unstained Control.fcs文件代表该样本没有进行任何增殖染色,可以作为背景噪音峰。请注意,FCS Express将背景峰拟合为增殖细胞的一个群体。之后我们会修改拟合参数,来告诉FCS Express将它作为背景。
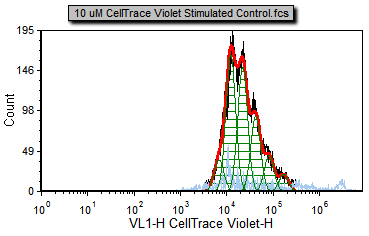 Figure T20.5a Proliferation positive control |
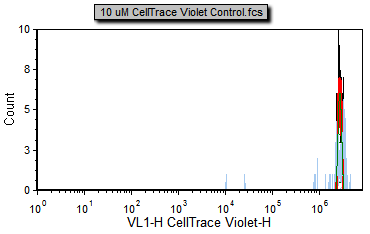 Figure T20.5b Proliferation negative control |
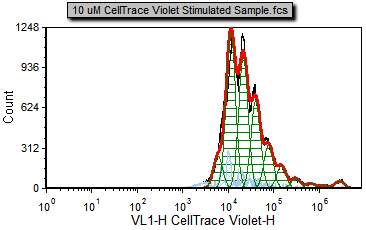 Figure T20.5c Proliferation stimulated sample |
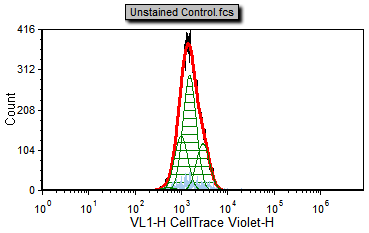 Figure T20.5d Proliferation unstained control |
您会注意到,不同的颜色会覆盖到不同的增殖样本上。细胞增殖曲线拟合的默认颜色如下:
黑色- 原始直方图轮廓。它包含所有的数据且并不代表拟合算法的输出,如果您将该数据展示在常规的柱状图上,也会看到相同的柱状图。
红色- 被拟合的数据的整体轮廓。该轮廓排除了算法认为是噪声的那部分信号。
浅蓝色- 噪声。这是算法判定为噪声信号的那部分,这部分信号不包括在统计计算中。
绿色- 模型拟合的增殖细胞群。这是对数据拟合后的实际曲线,代表父代细胞、未分裂细胞群和所有正在进行分裂的细胞代。
注意:增殖刺激样本中的细胞(图T20.5c)在10的6次方到10的7次方范围内,用浅蓝色显示,已通过模板证实为噪声信号。现在我们将修改Proliferation Fit Parameters(增殖拟合参数),来查看我们通过指定开始增殖位置来改善模型的拟合。注意:在门操作页,有一带有可见标识M1的柱状图。M1根据阴性对照来定义增殖的起始位置,我们将使用一个标记,来代表M1标识下数据的几何平均数,从而更准确的定义起始细胞代。
| 6. | 请使用Data(数据)标签→ Change Data on All(更改所以数据)→ Next/Previous(下一项/前一项)按钮来更改数据,直到到达10 uM CellTrace Violet Stimulated Control.fcs样本处。 |
| 7. | 右键点击Proliferation Data(增殖数据)页上的增殖绘图。 |
| 8. | 从弹出菜单中选择Format(格式)。 |
此时,Formatting(格式化)对话框将出现(图T20.6)。
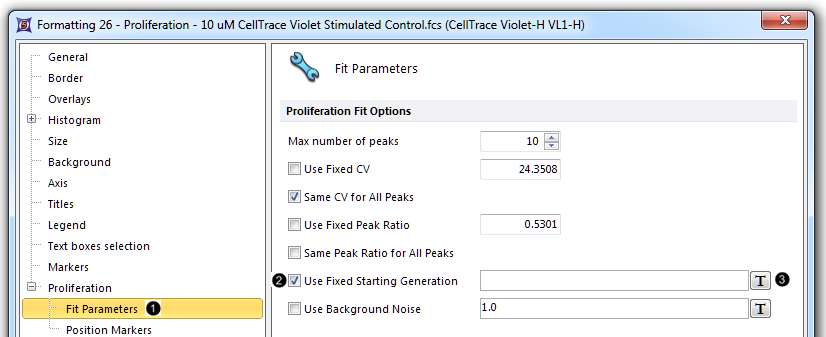
Figure T20.6 Adjusting proliferation fit parameters in the formatting dialog.
| 9. | 导航到Proliferation(增殖)→Fit Parameters(拟合参数)项目分类处(图T20.6 |
| 10. | 点击Use Fixed Starting Generation(使用固定起始细胞代)复选框(图T20.6 |
| 11. | 删除Use Fixed Starting Generation(使用固定起始细胞代)选项下,白色文本框中的数字(图T20.6)。 |
| 12. | 点击 |
| 此时,Insert a Token(插入一标记)对话框将出现。 |
| 13. | 双击Insert a Token(插入一标记)对话框中的Statistic(统计)选项。 |
| 14. | 选择绘图4 - Histogram作为Data Source (数据源),如下面的蓝色突出显示(图T20.7 |
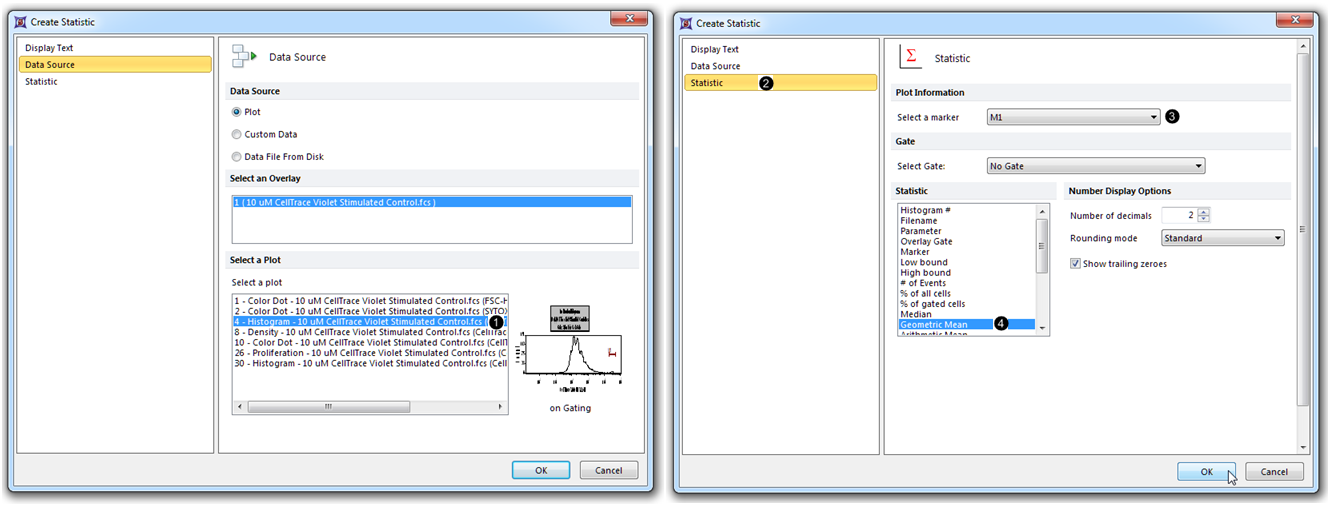
Figure T20.7 Adjusting proliferation fit parameters, continued.
| 15. | 点击左边的Statistic(统计)项目(图T20.7 |
| 16. | 设置Marker(标识)为M1(图T20.7 |
| 17. | 从Statistic(统计)列表中 选择GeometricMean(几何平均值)(图T20.7 |
| 18. | 点击OK(图T20.7,鼠标指针处)。 |
| 19. | 点击Proliferation(增殖)→Position Markers(位置标识)项目(图T20.8 |
| 20. | 选中复选框Show position marker on the plot(在绘图上显示位置标识),让Undivided cells peak(未分裂细胞峰)突出显示为蓝色 (图T20.8 |
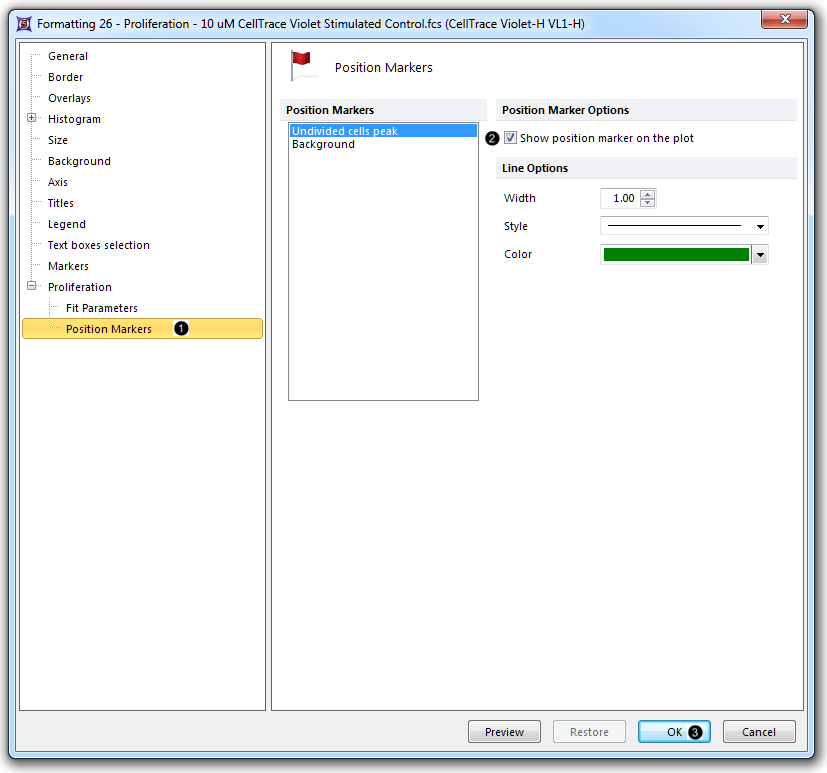
Figure T20.8 Checking the Show position marker on the plot box for the Undivided cells peak.
| 21. | 点击OK(图T20.8 |
现在,增殖柱状图看上去应如图T20.9所示。
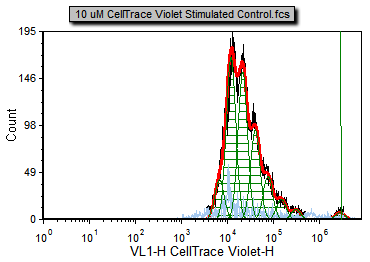
Figure T20.9 The proliferation plot after adjusting the starting generation and choosing to display the Undivided cells peak marker.
注意:在门操作页上,名为"Background Noise"的柱状图上,带有一相同名字的标识。在标识下方的区域,根据展示于柱状图上的Unstained Control.fcs样本,被定义作为背景噪音。为定义一起始细胞代,我们现在将应用和上面相同的步骤,来定义背景噪音。
| 22. | 右键点击Proliferation Data(增殖数据)页上的增殖绘图。 |
| 23. | 从弹出菜单选择Format(格式)。 |
此时,Formatting(格式化)对话框将出现,如图T20.6所示。
| 24. | 导航到Proliferation(增殖)→Fit Parameters(拟合参数)项目。 |
| 25. | 点击Use Background Noise(使用背景噪音)复选框。 |
| 26. | 删除Use Background Noise(使用背景噪音)选项下白色文本框中的数字。 |
| 27. | 点击 |
| 此时,Insert a Token(插入一个标记)对话框将出现。 |
| 28. | 双击Insert a Token(插入一个标记)对话框中的Statistic(统计)。 |
| 29. | 选择绘图30 - Histogram作为Data Source (数据源)。 |
| 30. | 点击左边的Statistic(统计)项目。 |
| 31. | 设置Marker(标识)为Background Noise(背景噪音)。 |
| 32. | 从Statistic(统计)列表中 选择GeometricMean(几何平均值)。 |
| 33. | 点击OK。 |
| 34. | 点击Proliferation(增殖)→Position Markers(位置标识)项目。 |
| 35. | 选中复选框Show position marker on the plot(在绘图上显示位置标识),让Background(背景)突出显示为蓝色。 |
| 36. | 点击OK。 |
现在增殖柱状图看上去应如图T20.10a所示,带有来自图T20.10b中柱状图的起始细胞代和背景噪音标识。
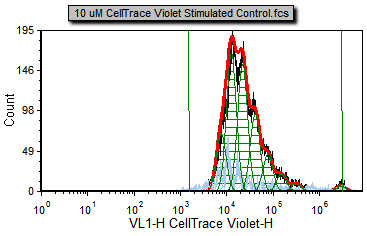 Figure T20.10a Proliferation plot with Background Noise Position and Starting Generation Markers defined. |
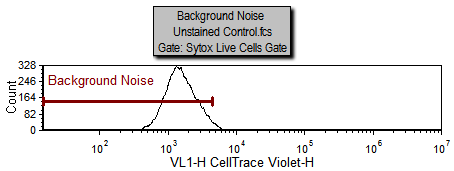
|
现在,拟合效果更好了,因为起始细胞代和背景噪音(在右边和左边,分别用绿色的竖线表示)是基于 M1或者Background Noise(背景噪音)标识中Sytox Live Cells(Sytox活细胞)门中的细胞来进行定义的。请注意,红色的曲线(拟合轮廓线)和黑色的曲线(原始数据轮廓线)考得更近了;R-Chi Square value(R-Chi平方值)更低了。虽然,针对更高端数据的拟合更好了,但您可以看见,很多曲线只做了轻微的改变。这是因为,在为起始细胞代添加更正确的标记值前,曲线拟合算法正确的找到了一个好的起始细胞代的近似值。然而,但在某些情况下,从对照样本中手动设置起始细胞代还是很重要的。另一方面,有时在增殖实验中,对照样本的信号峰的荧光值会和实验样本中的未分裂细胞的信号峰的荧光值有所偏差。有时,这些偏差可以让用户放弃使用对照柱状图,来设置未分裂细胞的位置。
您同样会注意到,如果您调整标识,让它扩大或者变小,增殖绘图拟合也会随之进行实时更新,因为起始细胞代和背景噪音是基于M1和Background noise(背景噪音)标识下的几何平均数统计的。关于计算拟合的描述,请参见下面的技术说明。
技术说明:以下是一个简短的描述拟合过程如何实现的技术注释。这些信息可以更好地帮助用户来理解,如何调整影响拟合的参数。如果不想理解这些细节,可以跳过。
|
下一章节,我们将对增殖柱状图进行格式化并显示统计。
